Технология секвенирования долго применялась для установления последовательности ДНК клеток одного вида (то есть «чистых культур»), а вот при попытке секвенировать материал из природных образцов возникало немало проблем. Пока не появилась метагеномика. Это направление занимается исследованием целых сообществ организмов, населяющих то или иное местообитание. Причем, оперируя метагеномными данными, ученые порой даже не имеют представления о внешнем виде и таксономической принадлежности изучаемых живых существ. Познакомимся с этой темой в деталях.
Начало
Собственно метагеномика стартовала с секвенирования отдельных ампликонов. И здесь, безусловно, самый известный маркер — ген 16s rRNA. По его последовательностям можно оценить разнообразие организмов сообщества. Так, благодаря этому методу в 1977 году ученые открыли новый домен жизни — Архей.
Но особенно преобразилась метагеномика с началом эры NGS. А именно стало распространяться полногеномное секвенирование и, соотвественно, Shotgun метагеномика. Она позволила определять таксономию и давать количественную оценку организмов вплоть до вида. Но главное ее отличие заключалось в том, что благодаря упорядочиванию множества различных частей генома каждого организма стало возможно выяснять еще и их функции в сообществе.
Шаг за шагом
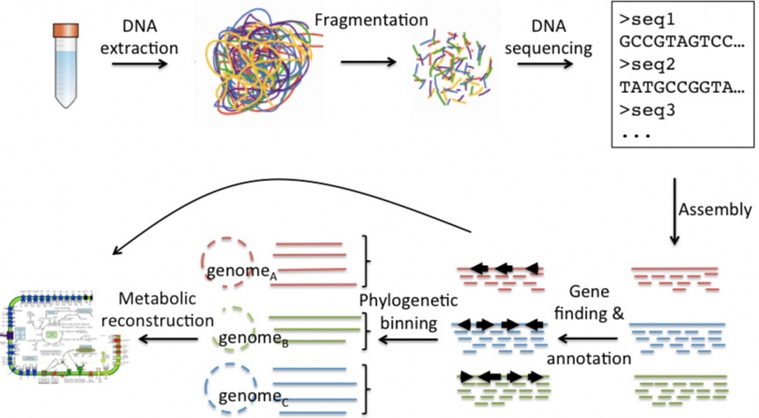
Какова же последовательность современного метагеномномного анализа? Сначала ДНК образца нарезается случайным образом, после чего фрагменты секвенируются. Затем их необходимо упорядочить и реконструировать в согласованную последовательность, уже по которой можно будет сделать вывод о видовом составе микроорганизмов в сообществе и их роли в нем.
Поскольку данные состоят из коротких считываний (ридов), необходим этап сборки, который создает длинные последовательности из коротких. Далее требуется сортировка «каши» из последовательностей по принадлежности к таксономическим группам — биннинг. Есть несколько способов определить, какому микроорганизму принадлежит последовательность. Например, это может быть анализ частоты использования кодонов, поскольку известно, что данная характеристика зависит от набора транспортных РНК и представляет собой уникальный «почерк» микроорганизма.
Другой вариант — анализ степени покрытия: широко представленные в сообществе микроорганизмы обладают широким покрытием, слабо представленные — узким. Вместе собираются контиги с одинаковым покрытием.
Кроме того, контиги можно собирать по сигнатурным наборам генов — это гены, которые содержаться в одной копии в любом геноме прокариот. В сборке должен быть полный набор этих единичных генов. По итогу сборки получаются MAGи. MAG — это геном, собранный с помощью метагенома. Он представляет собой сборку одного таксона.
В дальнейшем полученные MAGи нужно аннотировать. Как правило этот процесс начинается с автоаннотации: программа разбивает MAGи на белок-кодирующие последовательности и определяет входящие в их состав гены. Однако автоматика часто оперирует не самыми последними данными и требует дальнейшей ручной обработки или комбинации различных программ.
Выводы
Таким образом, мы выяснили, что shotgun метагеномика — это работающий инструмент, чтобы узнать больше о микробном сообществе. Однако она не лишена недостатков. В сообществе могут встречаться гены, чья функция до сих пор не установлена, что оставляет в общей картине белые пятна. Кроме того, наличие определенных генов ничего не говорит об их экспрессии, и для получения этой информации нужна уже метатранскриптомика.
Подготовлено при участии Анны Кузнецовой
