
Введение
Так как это мой первый пост на Хабре, поэтому напишу немного о себе.В 2018 окончил медицинский университет и в данный момент около 3.5 лет работаю патологоанатомом. Год назад после участия в проекте по аннотации гистологических изображений, заинтересовался digital pathology и начал самостоятельно изучать то, что с этим связано (Python, ML,DL на Coursera и DataCamp) и в частности computer vision.
Для получения опыта, решил самостоятельно сделать проект по классификации гистологических изображений что включало :
Получение полнослайдовых изображений (WSI) – подготовка датасета.
Аннотация изображений
Получение готового датасета (Train, Validation и Test)
Выбор и тренировка моделей
Тестирование моделей
Актуальность
Рак почки составляет около 2 % от всех диагнозов рака в мире.По данным GLOBOCAN диагностируется около 400 000 случаев в мире ежегодно (около 2.2% диагнозов рака).Соотношение мужчин и женщин около 2:1.(более детально по эпидемиологии можно почитать здесь ). Светлоклеточный рак почки - один из разновидностей рака, клетки которого имеют светлую ( отсюда и название) или эозинофильную (розовую) цитоплазму .Это самый частый вид почечноклеточного рака у взрослых и составляет 65-70 % от всех случаев. Часто связан с мутацией в гене - супрессоре опухолевого роста (VHL).
Датасет
Даже с учётом развития digital pathology и возможностью делиться слайдами, найти в открытом доступе достаточное количество изображений определенного вида опухоли (да и не только опухоли) пригодных для обучения модели и валидации довольно затруднительно.
Для проекта я выбрал коллекцию слайдов гистологических изображений, окрашенных гематоксилином и эозином, пациентов со светлоклеточным раком почки проекта Национального института рака США «Консорциум клинического протеомного анализа опухолей» (Clinical Proteomic Tumor Analysis Consortium, CPTAC) Clear Cell Renal Cell Carcinoma (CPTAC-CCRCC). Данные доступны на сайте Cancer Imaging Archive (TCIA).
Выбрал этот датасет из-за большого количества доступного материала (размер - 190 GB) в рамках одной патологии, из-за чего не должно было возникать проблем с подготовкой материала для тренировочного, валидационного и тестового наборов данных, обучением модели. Изображения доступны для скачивания в формате .svs.
Также присутствуют клинические данные пациентов, результаты генетических исследований (например статус VHL) . Для моей задачи они не были нужны, однако можно использовать в дальнейшем: например попытаться обучить модель предсказывающую статус гена VHL по гистологической картине.
Также хочу сразу отметить несколько минусов датасета :
Изображения без аннотаций. Так как я по образованию патологоанатом и исследование гистологического материала - это мои рабочие обязанности, то аннотировал слайды я самостоятельно.
Присутствуют слайды только хорошего качества : практически не встречаются артефакты и неинформативные изображения который не редки в обычной врачебной практике. Это влияет на результаты модели (результаты будут лучше, чем при использовании модели в реальной клинической практике).
Аннотация изображений
Как уже писал я ранее, аннотацию изображений проводил я самостоятельно с помощью программы QuPath 0.3.2.
Изначально для изображений я определил 6 классов :
Кровь (Blood)
Светлоклеточный рак почки (CCRCC)
Жир (Fat)
Ткань почки (Kidney)
Строма (Stroma) – соединительная ткань, которая не является специфическим элементом и может встречаться как в нормальной ткани, так и в опухоли. Сюда же входят стенки сосудов и др..
Артефакты (Artifact)
Однако из-за того, что практически все слайды хорошего качества, класс “Артефакты” я убрал.
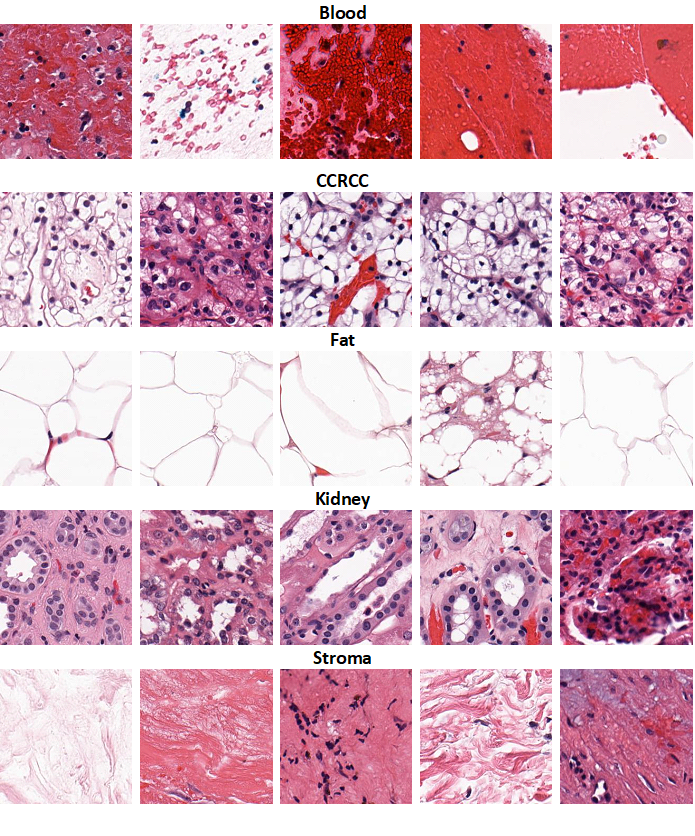
На слайдах с помощью программы QuPath проводилась аннотация изображений: размечались крупные зоны, относящиеся к какому-либо классу, а затем с помощью встроенного функционала программы эти зоны разделялись на фрагменты (Tiles), на которых в последующем и обучались нейронные сети.


Получение готового датасета (Train, Validation и Test)
Координаты объектов с метками классов экспортировались в формате JSON (Pretty JSON), а затем с помощью библиотеки OpenSlide на Python со слайдов извлекались изображения в формате .jpeg и размером 256х256 пикселей. К изображениям не применялись методы аугментации или нормализации окраски.
Весь датасет был разделён на 3 набора данных: Train, Validation и Test.
| Train (slides = 62, patients = 27) | Validation (slides = 16, patients = 7) | Test (slides = 52, patients = 15 |
Blood | 7697 | 2075 | 2446 |
CCRCC | 88429 | 16644 | 43758 |
Fat | 10924 | 10291 | 13062 |
Kidney | 50306 | 14885 | 42635 |
Stroma | 12150 | 2010 | 5739 |
Total | 169506 | 45905 | 107640 |

Отмечу, что слайды (а в последствии изображения) от одного и того же пациента могли присутствовать только в одном из наборов т.е. изображения от одного пациента не могли быть, например, в Train и Validation.
Нужно добавить, что c учётом того, что проект тренировочный и модели не будут использоваться в реальной клинической практике, из всех доступных полнослайдовых изображений для обучения и тестирования я использовал только часть. Это сделано естественно для удобства и экономии времени, чтобы иметь возможность более-менее адекватно обучить нейросети на Google Colab.
Выбор и обучение моделей глубокого обучения
Для обучения использовалось три готовых модели имплементированных на Keras:
InceptionV3
EfficientNetV2S
ResNet101V2
Модель | Количество параметров | Глубина | Top-1 accuracy на ImageNet | Top-5 accuracy на ImageNet |
InceptionV3 | 23.9 M | 189 | 77.9% | 93.7% |
EfficientV2S | 21.6 M | - | 83.9% | 96.7% |
ResNet101V2 | 44.7 M | 205 | 77.2% | 93.8% |
Функция потерь - "Categorical crossentropy"
Метрики : Accuracy score, ROC_AUC, PR_AUC, Precision, Recall
Результаты
Model | Set | Loss | Accuracy | ROC_AUC | PR_AUC | Precision | Recall |
Inception V3
| Train | 0.0541 | 0.9805 | 0.9989 | 0.9959 | 0.9814 | 0.9798 |
Validation | 0.3529 | 0.9248 | 0.9861 | 0.9271 | 0.9266 | 0.9233 | |
Test | 0.6850 | 0.8344 | 0.9650 | 0.8977 | 0.8405 | 0.8293 | |
EfficientNetV2S
| Train | 0.0824 | 0.9714 | 0.9975 | 0.9906 | 0.9731 | 0.9700 |
Validation | 0.3346 | 0.9280 | 0.9877 | 0.9154 | 0.9304 | 0.9263 | |
Test | 0.3971 | 0.9071 | 0.9834 | 0.9182 | 0.9107 | 0.9038 | |
ResNet101V2
| Train | 0.0584 | 0.9796 | 0.9984 | 0.9937 | 0.9807 | 0.9785 |
Validation | 0.2530 | 0.9390 | 0.9905 | 0.8920 | 0.9415 | 0.9370 | |
Test | 0.2264 | 0.9392 | 0.9874 | 0.9260 | 0.9419 | 0.9367 |
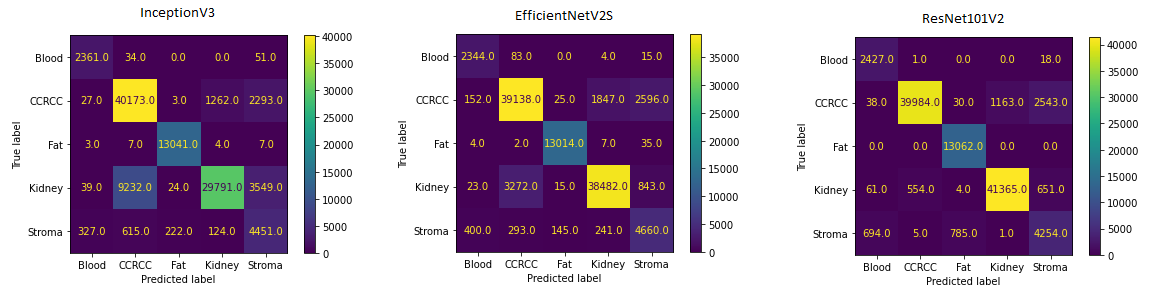
Наилучшие результаты как на валидационном, так и на тестовом наборе данных показала модель ResNet101V2 с точностью в 93.92 % и PR_AUC - 0.926. С классификацией изображений, содержащих жировую ткань или кровь трудностей у моделей не возникало. Довольно большое количество ошибок присутствует среди классов "Stroma", "CCRCC" и "Kidney".
"CCRCC" и "Kidney" : ткани имеют довольно сложную структуру, ткань почки, например, отличается в различных её отделах : присутствуют канальцы различного строения клубочки и др. Вероятно, для более точной классификации следуют использовать большее количество данных для обучения, что данный датасет позволяет. "Stroma" также разнородный класс и к тому же один из наименее представленных. В него я включал соединительную, мышечную ткани, стенки сосудов, зоны гиалиноза. Этот класс, возможно, следует разделить на несколько и более чётко обозначить их. Отмечу, что строма, жир и кровь - это вполне нормальные ткани организма и изображения представителей этих классов можно набирать из различных датасетов гистологических изображений.
Также для увеличения качества модели можно использовать методы нормализации окраски (имеются готовые библиотеки на Python). Ведь один и тот же материал, окрашенный разными лаборантами может выглядеть по разному : например, может превалировать розовый, или фиолетовый цвет, вид ядерного хроматина и др..
Всем, кто прочитал это спасибо за внимание :-) Буду рад всем советам касательно обучения моделей для компьютерного зрения в патологической анатомии или критике работы. Повторюсь, что не являюсь ML-инженером и данный проект исключительно любительский.
