Что-то давно не писал, вот решил написать промежуточную статью о развитии RNAInSpace. Первый этап статей собран в Получена траектория сворачивания вироидного рибозима или новости с фронтов при использовании ПО RNAInSpace. Попробуем начать второй этап.
Второй этап я собирался начать со сворачивания тРНК. Тут оказались некоторые проблемы. С другой стороны, есть интересный алгоритм CRA, который должен помочь решить мне эти проблемы. Он сложный и я его не понимаю. Но он реализован в некоторых ПО в основном для Linux. Что есть большое фи. В общем обо всем по порядку.
P.S. Ищу тех кто понимает математику и сможет помочь мне разобраться с алгоритмом CRA. С другой стороны, нуждаюсь в помощи тех кто использовал Gromacs.
Меня просили взять уже известную структуру тРНК, чтобы можно было сравнить модельную структуру с реальной. Сразу скажу, результатов пока полноценных нет. Но в тоже время мне интересно, если кто нибудь проведет моделирование на другом известном программном обеспечении, и покажет результаты. На мой взгляд — результатов не должно быть, сдается мне, что не одно известное ПО не свернет достаточно правильно тРНК.
Начнем разбираться с вторичной структуры (см. рисунок), которая дополнена межспиральными неканоническими водородными связями (они проставлены на основании литературы, и измерения связей в .pdb файле, который содержит реальную структуру этой тРНК). В моем подходе утверждается, что если получится образовать эти водородные связи, и еще некоторые связи стэкинг взаимодействия (которые нужны по сути лишь в промежуточных стадия сворачивания) — то мы и получим достаточно близкую к реальной модельную структуру.

Можно видеть, что с тРНК все намного сложнее, чем в прошлой статье с рибозимом. Если в рибозиме было сравнительно сложно образовать связи между тремя нуклеотидами, для правильной стыковки двух спиралей, то тут для тРНК нужно, чтобы T-спираль четко обкрутилась нуклеотидами 44-48, с другой стороны 8-9, а затем правильно стыковалось бы со спиралью D.
Не буду вдаваться в подробности, но последовательность сворачивания тут не простая, и противоречит т.н. иерархическим моделям сворачивания, согласно которым вначале образуются спирали (вторичные структуры), а затем они стыкуются более плотно. Это не так. Если спирали уже сформируются, то возможность стыковки становится практически не возможной.
На данный момент, могу показать модель, где образованы все спирали, кроме конечной (1-7 + 66-72). Но стыковка спиралей T и D, при необходимости создать все водородные связи еще не полностью происходит.

Видно, что если бы получилось правильно стыковать, и закрутить оставшуюся спираль — то профиль тРНК соответствовал реальному. А уже на сколько, измеряя по RMSD — это уже существенно вторичный вопрос. Но именно так сравнивают теперешние ученные в этой области. Хотя, пока я изучал реальную модель .pdb этой РНК — в ней тоже нашел несколько ошибок. Поэтому она тоже не идеальна, хотя и получена биологическими методами. Поэтому измерению по RMSD с моделью — доверять надо осторожно.
Зачем он?
Можно, конечно, отдать еще несколько месяцев и добиться стыковки спиралей T и D полуавтоматически сворачивая с помощью моего ПО RNAInSpace, вмешиваясь там где человек видит, что сворачивание идет не так, и играясь с последовательностью сворачивания и параметрами колебания.
Но так мы не сможем автоматизировать. Конечно, сворачивание в отдельную спираль — сейчас уже у меня автоматизировано, но когда спирали стыкуются — одна спираль должна подходить под другую совершенно уникальным образом для конкретной нуклеотидной последовательности. Эти спирали должны друг к другу как бы притереться.
И тут возникает проблема, которую я ощущал уже давно, но не мог достаточно четко сформулировать. вот некоторые выдержки и ссылка:
В общем удивительно, но ни в работотехнике, ни в других связанных с кинематикой областях, такой алгоритм не разработали.
Я задался целью поискать и найти как решается нужная мне проблема. Оказывается есть такие алгоритмы, и разрабатываются давно, хотя практически используются недавно. На русском, по обычаю :) ничего даже близко нет… что несколько обидно.
Что он конкретно делает?
Называются такие алгоритмы — concerted rotation algorithm. Мне попалась статья про т.н. CRISP алгоритм, который сделан для того, чтобы улучшить CRA алгоритм. Реализация CRISP и CRA была сделана в ПО PHAISTOS is a Markov chain Monte Carlo framework for protein structure simulations..
Главная цепь РНК (и белков) состоит из атомов соединенных связями. Длину связей нельзя менять, а можно лишь крутить атомы. Соответственно, если мы крутим какой-то атом, то вынуждена вращается вся цепь после этого атома (на траектории сворачивания «моего» рибозима видно, что один конец зафиксирован, а второй двигается. Задача зафиксировать оба конца в нужных точках).
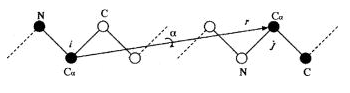
Но при сворачивании РНК, часто нужно зафиксировать атомы с двух сторон и уметь вращать лишь внутренний промежуток. Это я ранее и назвал «одной фундаментальной проблемой». Эти алгоритмы позволяют это сделать.
Это делается в два этапа. Первый — вращаем как хотим наш атом. Если мы не будем связанно вращать оставшийся конец, который зафиксирован, то произойдет разрыв цепи. Хорошо, мы идем на это. А вот вторым этапом, вращением последующего атома мы должны рассчитать так, чтобы разрыв полностью исчез. Как я понимаю там идет какая-та погрешность, и проблемы с тем, что такое закрытие разрыва может привести к пересечению атомов (связей) — и поэтому разные алгоритмы с разной эффективностью это делают.
Как это он делает?
А вот с этим и проблема. Может найдутся математики, которые мне тут помогут, я посмотрел на формулы, ужаснулся и думал плюнуть. По крайней мере, пока кто нибудь не поможет. Но за не имением своих знаний и математиков, и понимая важность данного алгоритма, я пошел смотреть готовые реализации.
Проблемы реализаций
Первая проблема — они написаны под Linux. Хороша новость, то что они написаны на Си. Вообще, смотря код писанный под Linux я никак не могу побороть у себя впечатление, что возвращаюсь в доисторическую эпоху. Говорить о совместимости с Windows (т.е. якобы многоплатформенности) — совершенно не приходится (а я как минимум 3 больших проекта «смотрел»).
Я даже не понимаю, почему люди хотят жить на 30 лет в прошлом. Но сейчас не об этом.
Второе, качество кода — Phaistos и Gromacs (о котором после) — не просто плохое, а ужасное. Плохо, это когда нет классов и объектов, или они переплетены со структурным стилем :), ужас — когда то, что есть написано так, что все переплетено.
Короче, найти отдельно реализованные методы сложно, приходится смотреть весь пакет, чтобы что-то понять. А это целые проекты по молекулярному моделированию.
Попробовал я партировать Phaistos в Windows… невозможно. Он использует библиотеку boost, где я после пару дней мучений пришел к однозначному выводу, библиотека сама по себе дрянь (использование шаблонов, в таком количестве, что код получается ужасным), и она не компилируется под Windows на MS Visual Studio. Забыли.
Далее мне повезло натолкнулся на другую реализацию метода CRA. Но тоже под здоровый проект Gromacs. Его партировать в Windows можно, и это практически получается. Только там надо разобраться с хитростями использования Gromacs. Может тут есть люди кто с ним работал?
Тогда возможно процесс у меня пойдет быстрее. По хорошему хочу сделать реализацию метода CRA под MS Visual Studio, как отдельную библиотеку, на вход подаешь координаты атомов, фиксированные атомы, желаемый поворот и — опля, алгоритм закрывает разрыв.
Второй этап я собирался начать со сворачивания тРНК. Тут оказались некоторые проблемы. С другой стороны, есть интересный алгоритм CRA, который должен помочь решить мне эти проблемы. Он сложный и я его не понимаю. Но он реализован в некоторых ПО в основном для Linux. Что есть большое фи. В общем обо всем по порядку.
P.S. Ищу тех кто понимает математику и сможет помочь мне разобраться с алгоритмом CRA. С другой стороны, нуждаюсь в помощи тех кто использовал Gromacs.
Сворачивание тРНК
Меня просили взять уже известную структуру тРНК, чтобы можно было сравнить модельную структуру с реальной. Сразу скажу, результатов пока полноценных нет. Но в тоже время мне интересно, если кто нибудь проведет моделирование на другом известном программном обеспечении, и покажет результаты. На мой взгляд — результатов не должно быть, сдается мне, что не одно известное ПО не свернет достаточно правильно тРНК.
Начнем разбираться с вторичной структуры (см. рисунок), которая дополнена межспиральными неканоническими водородными связями (они проставлены на основании литературы, и измерения связей в .pdb файле, который содержит реальную структуру этой тРНК). В моем подходе утверждается, что если получится образовать эти водородные связи, и еще некоторые связи стэкинг взаимодействия (которые нужны по сути лишь в промежуточных стадия сворачивания) — то мы и получим достаточно близкую к реальной модельную структуру.

Можно видеть, что с тРНК все намного сложнее, чем в прошлой статье с рибозимом. Если в рибозиме было сравнительно сложно образовать связи между тремя нуклеотидами, для правильной стыковки двух спиралей, то тут для тРНК нужно, чтобы T-спираль четко обкрутилась нуклеотидами 44-48, с другой стороны 8-9, а затем правильно стыковалось бы со спиралью D.
Не буду вдаваться в подробности, но последовательность сворачивания тут не простая, и противоречит т.н. иерархическим моделям сворачивания, согласно которым вначале образуются спирали (вторичные структуры), а затем они стыкуются более плотно. Это не так. Если спирали уже сформируются, то возможность стыковки становится практически не возможной.
На данный момент, могу показать модель, где образованы все спирали, кроме конечной (1-7 + 66-72). Но стыковка спиралей T и D, при необходимости создать все водородные связи еще не полностью происходит.

Видно, что если бы получилось правильно стыковать, и закрутить оставшуюся спираль — то профиль тРНК соответствовал реальному. А уже на сколько, измеряя по RMSD — это уже существенно вторичный вопрос. Но именно так сравнивают теперешние ученные в этой области. Хотя, пока я изучал реальную модель .pdb этой РНК — в ней тоже нашел несколько ошибок. Поэтому она тоже не идеальна, хотя и получена биологическими методами. Поэтому измерению по RMSD с моделью — доверять надо осторожно.
Метод CRA
Зачем он?
Можно, конечно, отдать еще несколько месяцев и добиться стыковки спиралей T и D полуавтоматически сворачивая с помощью моего ПО RNAInSpace, вмешиваясь там где человек видит, что сворачивание идет не так, и играясь с последовательностью сворачивания и параметрами колебания.
Но так мы не сможем автоматизировать. Конечно, сворачивание в отдельную спираль — сейчас уже у меня автоматизировано, но когда спирали стыкуются — одна спираль должна подходить под другую совершенно уникальным образом для конкретной нуклеотидной последовательности. Эти спирали должны друг к другу как бы притереться.
И тут возникает проблема, которую я ощущал уже давно, но не мог достаточно четко сформулировать. вот некоторые выдержки и ссылка:
Одна фундаментальная проблема — тут первые штрихи к описанию проблемы «обучения двух учителей» в данной задаче. А также там мы обсуждали возможность использования «инверсной кинематики» (которая используется для расчета суставов в робототехнике), но все это ужасно медленно. Соответственно разрабатываемый метод, может быть применен не только к проблеме сворачивания РНК, но и для улучшения решения проблем из области робототехники. Кстати, сворачивание сродни решению проблемы «парковки автомашины» в каждом отдельно взятом случае контакта двух нуклеотидов.
В общем удивительно, но ни в работотехнике, ни в других связанных с кинематикой областях, такой алгоритм не разработали.
Я задался целью поискать и найти как решается нужная мне проблема. Оказывается есть такие алгоритмы, и разрабатываются давно, хотя практически используются недавно. На русском, по обычаю :) ничего даже близко нет… что несколько обидно.
Что он конкретно делает?
Называются такие алгоритмы — concerted rotation algorithm. Мне попалась статья про т.н. CRISP алгоритм, который сделан для того, чтобы улучшить CRA алгоритм. Реализация CRISP и CRA была сделана в ПО PHAISTOS is a Markov chain Monte Carlo framework for protein structure simulations..
Главная цепь РНК (и белков) состоит из атомов соединенных связями. Длину связей нельзя менять, а можно лишь крутить атомы. Соответственно, если мы крутим какой-то атом, то вынуждена вращается вся цепь после этого атома (на траектории сворачивания «моего» рибозима видно, что один конец зафиксирован, а второй двигается. Задача зафиксировать оба конца в нужных точках).

Но при сворачивании РНК, часто нужно зафиксировать атомы с двух сторон и уметь вращать лишь внутренний промежуток. Это я ранее и назвал «одной фундаментальной проблемой». Эти алгоритмы позволяют это сделать.
Это делается в два этапа. Первый — вращаем как хотим наш атом. Если мы не будем связанно вращать оставшийся конец, который зафиксирован, то произойдет разрыв цепи. Хорошо, мы идем на это. А вот вторым этапом, вращением последующего атома мы должны рассчитать так, чтобы разрыв полностью исчез. Как я понимаю там идет какая-та погрешность, и проблемы с тем, что такое закрытие разрыва может привести к пересечению атомов (связей) — и поэтому разные алгоритмы с разной эффективностью это делают.
Как это он делает?
А вот с этим и проблема. Может найдутся математики, которые мне тут помогут, я посмотрел на формулы, ужаснулся и думал плюнуть. По крайней мере, пока кто нибудь не поможет. Но за не имением своих знаний и математиков, и понимая важность данного алгоритма, я пошел смотреть готовые реализации.
Проблемы реализаций
Первая проблема — они написаны под Linux. Хороша новость, то что они написаны на Си. Вообще, смотря код писанный под Linux я никак не могу побороть у себя впечатление, что возвращаюсь в доисторическую эпоху. Говорить о совместимости с Windows (т.е. якобы многоплатформенности) — совершенно не приходится (а я как минимум 3 больших проекта «смотрел»).
Я даже не понимаю, почему люди хотят жить на 30 лет в прошлом. Но сейчас не об этом.
Второе, качество кода — Phaistos и Gromacs (о котором после) — не просто плохое, а ужасное. Плохо, это когда нет классов и объектов, или они переплетены со структурным стилем :), ужас — когда то, что есть написано так, что все переплетено.
Короче, найти отдельно реализованные методы сложно, приходится смотреть весь пакет, чтобы что-то понять. А это целые проекты по молекулярному моделированию.
Попробовал я партировать Phaistos в Windows… невозможно. Он использует библиотеку boost, где я после пару дней мучений пришел к однозначному выводу, библиотека сама по себе дрянь (использование шаблонов, в таком количестве, что код получается ужасным), и она не компилируется под Windows на MS Visual Studio. Забыли.
Далее мне повезло натолкнулся на другую реализацию метода CRA. Но тоже под здоровый проект Gromacs. Его партировать в Windows можно, и это практически получается. Только там надо разобраться с хитростями использования Gromacs. Может тут есть люди кто с ним работал?
Тогда возможно процесс у меня пойдет быстрее. По хорошему хочу сделать реализацию метода CRA под MS Visual Studio, как отдельную библиотеку, на вход подаешь координаты атомов, фиксированные атомы, желаемый поворот и — опля, алгоритм закрывает разрыв.