Содержание
Основной смысл использования метрики Махаланобиса
1. Термины и определения
2. Расстояние Махаланобиса между двумя точками и между точкой и классом
2.1. Теоретические сведения
2.2. Алгоритм вычисления расстояния между двумя точками и между точкой и классом
2.3. Пример вычисления расстояния между двумя точками и между точкой и классом
3. Расстояние Махаланобиса между двумя классами
3.1. Теоретические сведения
3.2. Алгоритм вычисления расстояния между двумя классами
3.3. Пример вычисления расстояния между двумя классами
4. Расстояние Махаланобиса и метод k-ближайших соседей
5. Взвешенное расстояние Махаланобиса
6. Заключение
Если есть замечания или ошибки, пишите на почту quwarm@gmail.com или в комментариях.
Основной смысл использования расстояния Махаланобиса
На рисунке 1 два наблюдения изображены в виде красных точек.
Центр класса изображен в виде синей точки.

Вопрос — какое наблюдение ближе к центру класса?
Ответ зависит от того, как измеряется расстояние.
Если измерять расстояние по метрике Евклида, то получим, что расстояние от центра класса до точки
равно
, до точки
равно
, т. е. точка
ближе к центру класса.
Однако для этого распределения дисперсия в направлении меньше, чем дисперсия в направлении
, поэтому в некотором смысле точка
находится «на большем стандартном отклонении» от центра класса, чем
.
Эллипсы прогноза, изображенные на рисунке, подсказывают, что точка ближе по распределению, чем точка
. Измерив расстояние по Махаланобису, получим, что расстояние от центра класса
до точки
примерно равно
, до точки
примерно равно
, т. е. точка
ближе к центру класса. В этом и заключается основной смысл использования метрики Махаланобиса — учитывание дисперсий и ковариаций.
Кроме того, расстояние Махаланобиса предполагает, что точки множества эллипсоидально (частный случай — сферически) распределены вокруг центра масс.
1. Термины и определения
Метрика — функция, определяющая расстояние между любыми точками в метрическом пространстве , где
— размерность пространства.
Класс — конечное неупорядоченное множество схожих по некоторым критериям оптимальности точек:
, где
— количество точек в классе
.
Точка — конечное упорядоченное множество
значений признаков:
.
Будем обозначать буквой число признаков, а буквой
—
признак.
Примечания
Под словом «точка» подразумевается точка координатного -мерного пространства признаков. Причем замеченные сходства и различия между точками находятся в соответствии с расстояниями между ними.
Классы и точки в статье обозначаются как вектор-строки. В литературе и Интернете они иногда обозначаются как вектор-столбцы — тогда в некоторых частях формул операция транспонирования убирается, а в других добавляется.
В примерах: признак точки
из
класса обозначается как
.
Подразумевается, что одинаковых точек в классе (-ах) нет.
2. Расстояние Махаланобиса между двумя точками и между точкой и классом
Этот пункт включает внутриклассовое расстояние (расстояние между двумя точками из одного класса) и расстояние между точкой (не принадлежащей ни одному из классов) и классом.
2.1 Теоретические сведения
Расстояние Махаланобиса между двумя точками — мера расстояния между двумя случайными точками и
, одна из которых может (или обе могут) принадлежать некоторому классу
с матрицей ковариаций
:
Символ означает операцию транспонирования, а под
подразумевается матрица, обратная ковариационной.
Если матрица ковариаций является единичной матрицей, то расстояние Махаланобиса становится равным расстоянию Евклида.
Иначе говоря, если класс представляет собой упорядоченное множество нормированных (дисперсии равны 1) независимых (ковариации равны 0) точек, то расстояние Махаланобиса равно расстоянию Евклида.
Расстояние Махаланобиса безразмерно и масштабно-инвариантно.
Расстояние Махаланобиса является метрикой (доказательство здесь [internet archive] и здесь), т. е. между двумя точками
и
с матрицей ковариаций
в пространстве признаков удовлетворяет следующим аксиомам:
1. Аксиома тождества: ;
2. Аксиома симметрии: ;
3. Аксиома треугольника: .
Из этих аксиом следует неотрицательность функции расстояния: .
Из аксиом следует, что значение под корнем не меньше 0, однако при расчетах с использованием неточных вещественных чисел рекомендуется предварительно ограничивать диапазон результата слева значением 0 (max(0.0, value)) во избежание NaN, которое появляется после взятия корня (функция sqrt или возведение в степень 0.5) близкого к 0 слева числа (например, ). Этот нюанс часто не замечается.
Чтобы найти внутриклассовое расстояние Махаланобиса, нужно следовать вышеприведенной формуле — вычислить матрицу ковариаций класса и затем само расстояние между двумя точками в нем.
Чтобы найти расстояние Махаланобиса между точкой (не принадлежащей ни одному из классов) и классом, нужно также следовать вышеприведенной формуле — вычислить матрицу ковариаций класса и затем расстояние между точкой (не принадлежащей ни одному из классов) и центроидом класса (т. н. «расстояние до центроида»).
Для решения задачи классификации тестовой точки, нужно найти матрицы ковариаций всех классов. Затем с помощью подсчета расстояний от заданной точки до каждого класса выбрать класс, до которого расстояние минимально.
Некоторые методы (такие, как метод -ближайших соседей, который будет рассмотрен в п. 4) подразумевают вычисление расстояний не до центроидов классов, а до всех точек всех классов.
Перед тем, как находить матрицу ковариаций, необходимо вычислить математические ожидания* точек класса по признакам.
*
На практике математическое ожидание обычно оценивается как среднее арифметическое:
где — среднее арифметическое точек класса
по
признаку,
— количество точек в классе
,
—
признак точки
.
Центроид класса
:
Ковариация — это численное выражение свойства ковариантности двух признаков точек.
Свойство ковариантности означает, что признаки имеют тенденцию изменяться совместно (ковариантно).
Ковариационная матрица состоит из ковариаций между всеми парами признаков. Если количество признаков равно , то ковариационная матрица — матрица размерности
, имеющая вид:
Элементы ковариационной матрицы — ковариации — для набора точек вычисляются по формуле (несмещенная ковариация, англ. «sample covariance»):
где и
— математические ожидания по
и
признакам точек соответственно,
— количество точек в классе
.
Формулу нужно использовать только в том случае, если математические ожидания генеральной совокупности
и
рассматриваемого класса неизвестны. Если же они известны, то формула имеет вид (смещенная ковариация, англ. «population covariance»):
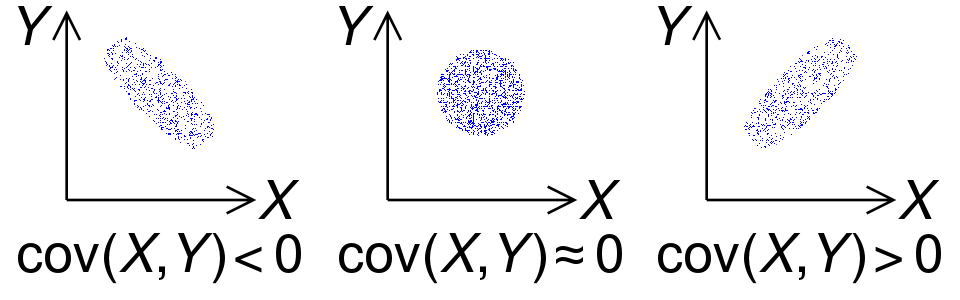
Ковариация обладает следующими важными свойствами:
Если при переходе от одной точки к другой
и
признаки увеличиваются (уменьшаются) вместе, то
;
Если при переходе от одной точки к другой
признак увеличивается, а
уменьшается (или наоборот), то
;
Если при переходе от одной точки к другой
и
признаки изменяются независимо, то
(обратное утверждение в общем случае неверно*).
Ковариация симметрична:
.
Неравенство Коши — Буняковского:
.
*
Пусть — равномерно распределенная случайная величина в
и
.
и
зависимы, но:
где — математическое ожидание.
Первые три свойства ковариации проиллюстрированы на рисунке 2.
Так как для вычисления метрики Махаланобиса требуется найти обратную к матрицу, а матрица обратима тогда и только тогда, когда она является квадратной и невырожденной (определитель не равен нулю), то необходимо и достаточно, чтобы определитель матрицы
не равнялся нулю. Однако такое требование является серьезным ограничением.
Известно, что ковариационная матрица необратима в следующих частных случаях:
1. Если по какому-либо признаку все точки класса имеют одно и то же значение и, следовательно, среднеквадратическое отклонение по признаку
равно нулю.
Пример: .
2. Если ковариации всех признаков максимальны (, «perfect covariance»). Примеры:
— идеальная положительная ковариация;
— идеальная отрицательная ковариация.
3. Если количество точек в классе меньше количества признаков
плюс
:
Есть и другие случаи.
Что делать, если ковариационная матрица необратима?
Единственно правильного подхода не существует.
Однако существует целая область исследований, направленная на регуляризацию этой проблемы.
Три приведенные выше и некоторые другие проблемы могут быть решены следующими способами:
1. Два способа для первого случая
Добавить больше точек в класс, чтобы среднеквадратическое отклонение (аналогично — дисперсия) по признаку
не равнялось нулю.
Убрать признак
из рассмотрения.
2. Метрика Евклида — Махаланобиса
Использовать модификацию метрики Махаланобиса (например, для второго случая), — метрику Евклида-Махаланобиса (из статьи):
где — единичная матрица того же размера, что и
.
Эта метрика устраняет недостаток метрики Махаланобиса, поскольку элементы её главной диагонали всегда больше нуля.
3. Псевдообратный подход
Помимо обратной матрицы существует псевдообратная матрица.
Операция — псевдообратное преобразование матрицы (обратное преобразование Мура — Пенроуза).
Функции вычисления псевдообратной матрицы:
— ginv в библиотеке MASS (R);
— pinv в библиотеке numpy (Python);
— pinv в MATLAB;
— pinv в Octave.
Псевдообратная матрица, обозначаемая , (в отрыве от темы статьи) определяется как матрица, которая «решает» задачу наименьших квадратов:
, где
— прямоугольная матрица, в которой число строк (уравнений) больше числа столбцов (переменных); такая система уравнений в общем случае не имеет решения, поэтому эту систему можно «решить» только в смысле выбора такого вектора
, чтобы минимизировать «расстояние» между векторами
и
.
Псевдообратная матрица может быть найдена с помощью сингулярного разложения матрицы. Причем для любой матрицы над вещественными числами существует псевдообратная матрица и притом только одна.
Также важно отметить тот факт, что если обратную матрицу можно найти (иначе говоря, исходная матрица
— квадратная и невырожденная), то псевдообратная будет с
совпадать:
.
Формула вычисления расстояния:
Псевдообратный подход иногда применяют в расстоянии Махаланобиса, но: «Мы получаем значительно меньшую точность классификации при использовании псевдообратных матриц. Действительно, псевдообратный подход генерирует вдвое больше ошибок, чем метод усадки ковариационной матрицы или метод диагональной матрицы» (из статьи).
Кроме того, далее будет продемонстрирован случай, когда при использовании псевдообратного подхода нарушается аксиома тождества (из-за чего этот подход называют псевдорасстоянием Махаланобиса или псевдометрикой).
4. Метод усадки ковариационной матрицы
Метод усадки (shrinkage) ковариационной матрицы — это метод оценки задач с небольшим количеством точек и большим количеством признаков (т. е. для третьего случая).
Смысл этого метода в замене матрицы на матрицу
, где
— некоторая подходящая положительно определенная матрица,
— коэффициент усадки, причем наименьшее собственное значение матрицы
должно быть не меньше
, умноженной на наименьшее собственное значение
.
В расстоянии Махаланобиса:
Предложение Olivier Ledoit и Michael Wolf — , где
— сумма диагональных элементов матрицы
, деленная на число признаков,
— единичная матрица, а
вычисляется в соответствии с приведенным авторами алгоритмом.
Реализация алгоритма, предложенного авторами, на Python имеется в библиотеке scikit-learn (sklearn.covariance.LedoitWolf, sklearn.covariance.ledoit_wolf, sklearn.covariance.ledoit_wolf_shrinkage).
На стр. 8 написано, что «в отличие от псевдообратного подхода, метод усадки ковариационной матрицы генерирует обобщенную меру расстояния, которая является метрикой» (адаптированный перевод). Это утверждение может ввести в заблуждение в отрыве от контекста — три перечисленных выше условия (про , про
, про собственные значения) обязательны, иначе результат может быть неверным.
Следующий пример демонстрирует несоблюдение условия .
Пусть , тогда в соответствии с предложением в этой и этой статьях (реализация на Python):
— ;
— расстояние от точки до точки
:
;
— расстояние от точки до точки
:
;
— расстояние от точки до точки
:
;
— расстояние от точки до точки
:
.
В данном случае предложение:
где — диагональная матрица со значениями на диагонали
.
Также есть Shrunk Covariance (sklearn.covariance.ShrunkCovariance, sklearn.covariance.shrunk_covariance). Однако он не находит , а предлагает пользовательский выбор (по умолчанию
).
Матрица (как и в предложении Ledoit — Wolf): .
Общую информацию об усадке можно почитать в википедии.
Причем стоит обратить внимание на то, что LedoitWolf и ShrunkCovariance (и некоторые другие методы) используют empirical_covariance, которая вычисляет смещенную ковариацию (англ. «population covariance», формула ).
5. Нормализованное расстояние Евклида
Если матрица ковариаций диагональная (но необязательно единичная), то получившаяся мера расстояния носит название «нормализованное расстояние Евклида»:
где — среднеквадратическое отклонение точек класса (к которому относится точка
и/или точка
) по
признаку (исправленное СКО, «corrected sample standard deviation»):
где — математическое ожидание точек класса к которому относится точка
и/или точка
) по
признаку.
В случае среднего арифметического:
Формулу нужно использовать только в том случае, если математические ожидания генеральной совокупности
рассматриваемого класса неизвестны. Если же они известны, то формула имеет вид (неисправленное СКО, англ. «uncorrected sample standard deviation»):
Это расстояние не учитывает какую-либо зависимость между признаками и требует не равные нулю среднеквадратические отклонения.
6. Метод диагональной матрицы
Из статьи:
Или более полно:
Это расстояние не учитывает какую-либо зависимость между признаками и требует не равные нулю среднеквадратические отклонения.
Есть и другие способы, но они выходят за рамки этой статьи.
Во всяком случае, как показывает практика, нужно использовать примерно в 10 раз больше точек, чем признаков. Ведь задача не только в том, чтобы ковариация была хорошо обусловлена, но также и в том, чтобы она была точной.
2.2 Алгоритм вычисления расстояния между двумя точками и между точкой и классом
Шаг 1. Вычислить математические ожидания значений признаков точек класса.
Шаг 2. Вычислить среднеквадратические отклонения значений признаков точек класса.
Шаг 3. Вычислить ковариации между всеми парами признаков точек класса и составить ковариационную матрицу.
Шаг 4. Если матрица обратима, то вычислить расстояние по Махаланобису. Если нет, то попробовать один из вышеперечисленных способов решения.
2.3 Пример вычисления расстояния между двумя точками и между точкой и классом
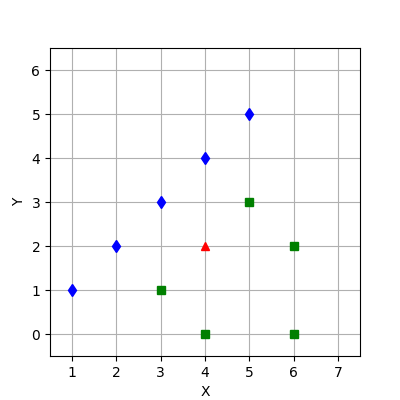
Пусть имеется тестовая точка и два следующих класса (рис. 3):
Шаг 1. Вычислим математические ожидания значений признаков точек классов.
Шаг 2. Вычислим среднеквадратические отклонения значений признаков точек классов.
По первому и второму признакам точек первого класса:
По первому и второму признакам точек второго класса:
Шаг 3. Вычислим ковариации между всеми парами признаков точек классов и составим ковариационные матрицы.
Для первого класса.
Получим следующую матрицу ковариаций:
Вычислим определитель этой матрицы: . Следовательно, матрица
необратима.
Для второго класса.
Получим следующую матрицу ковариаций:
Вычислим определитель этой матрицы: . Следовательно, матрица
обратима.
Код на Python 3.6 с использованием библиотеки numpy 1.19.5
import numpy as np
classes = [
np.array([[1, 1], [2, 2], [3, 3], [4, 4], [5, 5]]),
np.array([[3, 1], [4, 0], [6, 0], [6, 2], [5, 3]])
]
centroids = [class_.mean(axis=0) for class_ in classes]
standard_deviations = [class_.std(axis=0, ddof=1) for class_ in classes]
covariance_matrices = np.array([np.cov(class_, rowvar=False, ddof=1) for class_ in classes])
det_covariance_matrices = [np.linalg.det(cov) for cov in covariance_matrices]
print("Centroids:", *centroids)
print("Standard deviations:", *standard_deviations)
print("Covariance matrices:", *covariance_matrices.tolist())
print("Determinants of covariance matrices:", det_covariance_matrices)
Вывод:
Centroids: [3. 3.] [4.8 1.2]
Standard deviations: [1.58113883 1.58113883] [1.30384048 1.30384048]
Covariance matrices: [[2.5, 2.5], [2.5, 2.5]] [[1.7, 0.3], [0.3, 1.7]]
Determinants of covariance matrices: [0.0, 2.8]Шаг 4. Если матрица обратима, то вычислим расстояние по Махаланобису и расстояние по Евклиду — Махаланобису. Если матрица необратима, то попробуем несколько способов решения этой проблемы.
Различают расстояние, измеряемое по принципу ближайшего соседа, дальнего соседа и расстояние, измеряемое по принципу центроида.
Измерим расстояния между тестовой точкой и всеми точками классов, включая точку центроида.
Первый класс. Как уже было сказано ранее — для матрицы ковариаций первого класса нельзя найти обратную матрицу, поэтому расстояние между тестовой точкой и первым классом будем вычислять по 5 формулам: (1) метрика Евклида — Махаланобиса, (2) метод усадки ковариационной матрицы (LedoitWolf), (3) псевдообратный подход, (4) нормализованное расстояние Евклида, (5) метод диагональной матрицы — и выберем среди них наиболее правдоподобную:
1. Метрика Евклида — Махаланобиса.
Код на Python 3.6 с использованием библиотеки numpy 1.19.5
import numpy as np
def mahalanobis(point_from, point_to, inverse_covariance_matrix):
delta = point_from - point_to
return max(np.float64(0), np.dot(np.dot(delta, inverse_covariance_matrix), delta)) ** 0.5
test_point = np.array([4., 2.])
class_ = np.array([[1., 1.], [2., 2.], [3., 3.], [4., 4.], [5., 5.]])
covariance_matrix = np.cov(class_, rowvar=False, ddof=1)
inverse_covariance_matrix = np.linalg.inv(covariance_matrix + np.identity(covariance_matrix.shape[0]))
print("Обратная ковариационная матрица:\n", inverse_covariance_matrix, sep='')
for point_to in [class_.mean(axis=0), *class_]:
print("d_E-M (", test_point, ", ", point_to, ", (COV+E)^(-1)) = ",
mahalanobis(test_point, point_to, inverse_covariance_matrix), sep='')
Вывод:
Обратная ковариационная матрица:
[[ 0.58333333 -0.41666667]
[-0.41666667 0.58333333]]
d_E-M ([4. 2.], [3. 3.], (COV+E)^(-1)) = 1.414213562373095
d_E-M ([4. 2.], [1. 1.], (COV+E)^(-1)) = 1.8257418583505538
d_E-M ([4. 2.], [2. 2.], (COV+E)^(-1)) = 1.5275252316519465
d_E-M ([4. 2.], [3. 3.], (COV+E)^(-1)) = 1.414213562373095
d_E-M ([4. 2.], [4. 4.], (COV+E)^(-1)) = 1.5275252316519465
d_E-M ([4. 2.], [5. 5.], (COV+E)^(-1)) = 1.8257418583505536Первая точка — точка центроида, которая совпадает с одной из точек класса.
2. Метод усадки ковариационной матрицы (LedoitWolf).
Код на Python 3.6 с использованием библиотек numpy 1.19.5 и scikit-learn 0.24.1
import numpy as np
from sklearn.covariance import LedoitWolf
def mahalanobis(point_from, point_to, inverse_covariance_matrix):
delta = point_from - point_to
return max(np.float64(0), np.dot(np.dot(delta, inverse_covariance_matrix), delta)) ** 0.5
def approx(number, *, sign, epsilon=1e-4):
return number + np.sign(sign) * epsilon
test_point = np.array([4., 2.])
class_ = np.array([[1., 1.], [2., 2.], [3., 3.], [4., 4.], [5., 5.]])
lw = LedoitWolf().fit(class_)
lw_covariance_matrix = lw.covariance_
lw_lambda = lw.shrinkage_
covariance_matrix = np.cov(class_, rowvar=False, ddof=0)
mu = np.sum(np.trace(covariance_matrix)) / class_.shape[0]
T = mu * np.identity(class_.shape[1])
print("T:", *T)
print("COV(*):", *lw_covariance_matrix)
print("Lambda:", lw_lambda)
# Первое условие - T является положительно определенной матрицей
# (достаточное условие: все собственные значения матрицы T положительны)
# ddof=0, т. к. LedoitWolf вызывает empirical_covariance (исп. смещенную ковариацию)
first_condition = (np.linalg.eig(T)[0] > approx(0., sign=+1)).all()
print("All(", np.linalg.eig(T)[0], ") > 0 ? -> ", first_condition, sep='')
# Второе условие - лямбда в полуинтервале (0, 1]
second_condition = approx(0., sign=+1) < lw_lambda <= 1
print("Lambda =", lw_lambda, "in (0, 1] ? ->", second_condition)
# Третье условие - наименьшее собственное значение матрицы COV(*)
# должно быть не меньше lambda, умноженной на наименьшее собственное значение T
cov_eig = min(np.linalg.eig(lw_covariance_matrix)[0])
lambda_t_eig = lw_lambda * min(np.linalg.eig(T)[0])
third_condition = cov_eig >= lambda_t_eig
print(cov_eig, ">=", lambda_t_eig, "? ->", third_condition)
conditions = [first_condition, second_condition, third_condition]
if all(conditions):
print("Все три условия выполнены")
# Обратная матрица
inverse_lw_covariance_matrix = np.linalg.inv(lw_covariance_matrix)
print("Обратная ковариационная матрица:\n", inverse_lw_covariance_matrix, sep='')
for point_to in [class_.mean(axis=0), *class_]:
print("d_M(*) (", test_point, ", ", point_to, ", COV(*)) = ",
mahalanobis(test_point, point_to, inverse_lw_covariance_matrix), sep='')
else:
print("Невыполненные условия (1-3): ", [i for i, x in enumerate(conditions, 1) if not x])
Вывод:
T: [0.8 0. ] [0. 0.8]
COV(*): [2. 1.44] [1.44 2. ]
Lambda: 0.27999999999999997
All([0.8 0.8]) > 0 ? -> True
Lambda = 0.27999999999999997 in (0, 1] ? -> True
0.56 >= 0.22399999999999998 ? -> True
Все три условия выполнены
Обратная ковариационная матрица:
[[ 1.03820598 -0.74750831]
[-0.74750831 1.03820598]]
d_M(*) ([4. 2.], [3. 3.], COV(*)) = 1.889822365046136
d_M(*) ([4. 2.], [1. 1.], COV(*)) = 2.4283759936997833
d_M(*) ([4. 2.], [2. 2.], COV(*)) = 2.037847864848056
d_M(*) ([4. 2.], [3. 3.], COV(*)) = 1.889822365046136
d_M(*) ([4. 2.], [4. 4.], COV(*)) = 2.037847864848056
d_M(*) ([4. 2.], [5. 5.], COV(*)) = 2.4283759936997833Первая точка — точка центроида, которая совпадает с одной из точек класса.
3. Псевдообратный подход.
Ранее уже было сказано про псевдообратные матрицы. Их недостаток использования демонстрируется в следующем примере.
Как видим, нарушена аксиома тождества — расстояние между двумя различными точками равно нулю.
Код на Python 3.6 с использованием библиотеки numpy 1.19.5
import numpy as np
def mahalanobis(point_from, point_to, inverse_covariance_matrix):
delta = point_from - point_to
return max(np.float64(0), np.dot(np.dot(delta, inverse_covariance_matrix), delta)) ** 0.5
test_point = np.array([4., 2.])
class_ = np.array([[1., 1.], [2., 2.], [3., 3.], [4., 4.], [5., 5.]])
covariance_matrix = np.cov(class_, rowvar=False, ddof=1)
# Используется сингулярное разложение (Singular Value Decomposition, SVD)
# для вычисления псевдообратной матрицы
pseudo_inverse_covariance_matrix = np.linalg.pinv(covariance_matrix)
print("Псевдообратная ковариационная матрица:\n", pseudo_inverse_covariance_matrix, sep='')
for point_to in [class_.mean(axis=0), *class_]:
print("d_M+ (", test_point, ", ", point_to, ", COV+) = ",
mahalanobis(test_point, point_to, pseudo_inverse_covariance_matrix), sep='')
Вывод:
Псевдообратная ковариационная матрица:
[[0.1 0.1]
[0.1 0.1]]
d_M+ ([4. 2.], [3. 3.], COV+) = 0.0
d_M+ ([4. 2.], [1. 1.], COV+) = 1.2649110640673513
d_M+ ([4. 2.], [2. 2.], COV+) = 0.6324555320336757
d_M+ ([4. 2.], [3. 3.], COV+) = 0.0
d_M+ ([4. 2.], [4. 4.], COV+) = 0.6324555320336757
d_M+ ([4. 2.], [5. 5.], COV+) = 1.2649110640673513Первая точка — точка центроида, которая совпадает с одной из точек класса.
4. Нормализованное расстояние Евклида.
Код на Python 3.6 с использованием библиотеки numpy 1.19.5
import numpy as np
def euclid_std(point_from, point_to, standard_deviations):
return sum(((point_from - point_to) / standard_deviations) ** 2) ** 0.5
def approx(number, *, sign, epsilon=1e-4):
return number + np.sign(sign) * epsilon
test_point = np.array([4., 2.])
class_ = np.array([[1., 1.], [2., 2.], [3., 3.], [4., 4.], [5., 5.]])
standard_deviations = class_.std(axis=0, ddof=1)
# Если не близко и не равно 0
std_le_0 = standard_deviations <= approx(0., sign=+1, epsilon=1e-6)
print("Среднеквадратические отклонения:\n", standard_deviations, sep='')
if std_le_0.any():
print("СКО по следующим признакам равно 0: ", np.where(std_le_0)[0])
else:
for point_to in [class_.mean(axis=0), *class_]:
print("d_std (", test_point, ", ", point_to, ", sigma) = ",
euclid_std(test_point, point_to, standard_deviations), sep='')
Вывод:
Среднеквадратические отклонения:
[1.58113883 1.58113883]
d_std ([4. 2.], [3. 3.], sigma) = 0.8944271909999159
d_std ([4. 2.], [1. 1.], sigma) = 1.9999999999999998
d_std ([4. 2.], [2. 2.], sigma) = 1.2649110640673518
d_std ([4. 2.], [3. 3.], sigma) = 0.8944271909999159
d_std ([4. 2.], [4. 4.], sigma) = 1.2649110640673518
d_std ([4. 2.], [5. 5.], sigma) = 1.9999999999999998Первая точка — точка центроида, которая совпадает с одной из точек класса.
5. Метод диагональной матрицы.
Код на Python 3.6 с использованием библиотеки numpy 1.19.5
import numpy as np
def euclid_std(point_from, point_to, standard_deviations):
return sum(((point_from - point_to) / standard_deviations) ** 2) ** 0.5
def euclid_diag(point_from, point_to, standard_deviations):
return euclid_std(point_from, point_to, standard_deviations) \
* (np.prod(standard_deviations ** 2)) ** (1. / point_from.shape[0])
def approx(number, *, sign, epsilon=1e-4):
return number + np.sign(sign) * epsilon
test_point = np.array([4., 2.])
class_ = np.array([[1., 1.], [2., 2.], [3., 3.], [4., 4.], [5., 5.]])
standard_deviations = class_.std(axis=0, ddof=1)
# Если не близко и не равно 0
std_le_0 = standard_deviations <= approx(0., sign=+1, epsilon=1e-6)
print("Среднеквадратические отклонения:\n", standard_deviations, sep='')
if std_le_0.any():
print("СКО по следующим признакам равно 0: ", np.where(std_le_0)[0])
else:
for point_to in [class_.mean(axis=0), *class_]:
print("d_diag (", test_point, ", ", point_to, ", sigma) = ",
euclid_diag(test_point, point_to, standard_deviations), sep='')
Вывод:
Среднеквадратические отклонения:
[1.58113883 1.58113883]
d_diag ([4. 2.], [3. 3.], sigma) = 2.2360679774997902
d_diag ([4. 2.], [1. 1.], sigma) = 5.0
d_diag ([4. 2.], [2. 2.], sigma) = 3.16227766016838
d_diag ([4. 2.], [3. 3.], sigma) = 2.2360679774997902
d_diag ([4. 2.], [4. 4.], sigma) = 3.16227766016838
d_diag ([4. 2.], [5. 5.], sigma) = 5.0Первая точка — точка центроида, которая совпадает с одной из точек класса.
Второй класс. Для матрицы ковариаций второго класса можно найти обратную матрицу (это можно сделать несколькими способами). Вручную легче всего вычислять при помощи матрицы алгебраических дополнений:
где — определитель матрицы
,
— транспонированная матрица алгебраических дополнений для матрицы ковариаций второго класса.
Код на Python 3.6 с использованием библиотеки numpy 1.19.5
import numpy as np
def mahalanobis(point_from, point_to, inverse_covariance_matrix):
delta = point_from - point_to
return max(np.float64(0), np.dot(np.dot(delta, inverse_covariance_matrix), delta)) ** 0.5
def approx(number, *, sign, epsilon=1e-4):
return number + np.sign(sign) * epsilon
test_point = np.array([4., 2.])
class_ = np.array([[3., 1.], [4., 0.], [6., 0.], [6., 2.], [5., 3.]])
covariance_matrix = np.cov(class_, rowvar=False, ddof=1)
if abs(np.linalg.det(covariance_matrix)) <= approx(0., sign=+1):
print("Определитель равен 0. Матрица необратима.")
else:
inverse_covariance_matrix = np.linalg.inv(covariance_matrix)
print("Обратная ковариационная матрица (d_M):\n", inverse_covariance_matrix, sep='')
for point_to in [class_.mean(axis=0), *class_]:
print("d_M (", test_point, ", ", point_to, ", COV^(-1)) = ",
mahalanobis(test_point, point_to, inverse_covariance_matrix), sep='')
covariance_matrix = covariance_matrix + np.identity(class_.shape[1])
inverse_covariance_matrix = np.linalg.inv(covariance_matrix)
print("Обратная ковариационная матрица (d_E-M):\n", inverse_covariance_matrix, sep='')
for point_to in [class_.mean(axis=0), *class_]:
print("d_E-M (", test_point, ", ", point_to, ", (COV+E)^(-1)) = ",
mahalanobis(test_point, point_to, inverse_covariance_matrix), sep='')
Вывод:
Обратная ковариационная матрица (d_M):
[[ 0.60714286 -0.10714286]
[-0.10714286 0.60714286]]
d_M ([4. 2.], [4.8 1.2], COV^(-1)) = 0.9561828874675149
d_M ([4. 2.], [3. 1.], COV^(-1)) = 1.0
d_M ([4. 2.], [4. 0.], COV^(-1)) = 1.5583874449479593
d_M ([4. 2.], [6. 0.], COV^(-1)) = 2.3904572186687876
d_M ([4. 2.], [6. 2.], COV^(-1)) = 1.5583874449479593
d_M ([4. 2.], [5. 3.], COV^(-1)) = 1.0
Обратная ковариационная матрица (d_E-M):
[[ 0.375 -0.04166667]
[-0.04166667 0.375 ]]
d_E-M ([4. 2.], [4.8 1.2], (COV+E)^(-1)) = 0.7302967433402214
d_E-M ([4. 2.], [3. 1.], (COV+E)^(-1)) = 0.8164965809277259
d_E-M ([4. 2.], [4. 0.], (COV+E)^(-1)) = 1.224744871391589
d_E-M ([4. 2.], [6. 0.], (COV+E)^(-1)) = 1.8257418583505536
d_E-M ([4. 2.], [6. 2.], (COV+E)^(-1)) = 1.224744871391589
d_E-M ([4. 2.], [5. 3.], (COV+E)^(-1)) = 0.8164965809277259Первая точка — точка центроида.
• По принципу ближнего соседа
Первый класс:
1. Метрика Евклида — Махаланобиса: ;
2. Метод усадки ковариационной матрицы (LedoitWolf): ;
3. Псевдообратный подход: ;
4. Нормализованное расстояние Евклида: ;
5. Метод диагональной матрицы: .
Второй класс (метрика Махаланобиса): .
Второй класс (метрика Евклида — Махаланобиса): .
Судя по рисунку 3, более правдоподобны (для данного примера и принципа) метрика Евклида — Махаланобиса, метод усадки ковариационной матрицы и метод диагональной матрицы.
• По принципу дальнего соседа
Первый класс:
1. Метрика Евклида — Махаланобиса: ;
2. Метод усадки ковариационной матрицы (LedoitWolf): ;
3. Псевдообратный подход: ;
4. Нормализованное расстояние Евклида: ;
5. Метод диагональной матрицы: .
Второй класс (метрика Махаланобиса): .
Второй класс (метрика Евклида — Махаланобиса): .
Судя по рисунку 3, более правдоподобны (для данного примера и принципа) метод усадки ковариационной матрицы и метод диагональной матрицы.
• По принципу центроида
Первый класс:
1. Метрика Евклида — Махаланобиса: ;
2. Метод усадки ковариационной матрицы (LedoitWolf): ;
3. Псевдообратный подход: ;
4. Нормализованное расстояние Евклида: ;
5. Метод диагональной матрицы: .
Второй класс (метрика Махаланобиса): .
Второй класс (метрика Евклида — Махаланобиса): .
Судя по рисунку 3, более правдоподобны (для данного примера и принципа) метрика Евклида — Махаланобиса, метод усадки ковариационной матрицы и метод диагональной матрицы.
Результат
Наиболее правдоподобными методами для данного примера являются метод усадки ковариационной матрицы и метод диагональной матрицы.
3. Расстояние Махаланобиса между двумя классами
Этот пункт включает расстояние между двумя классами и расстояние между точкой (представляющей единственный объект класса) и классом.
Расстояние Махаланобиса между двумя классами является квазиметрикой, т. е.:
— удовлетворяет условиям ,
,
.
— не удовлетворяет условию (в общем случае) .
Подробнее здесь.
3.1 Теоретические сведения
Расстояние Махаланобиса между двумя классами — мера расстояния между двумя классами и
с центроидами
и
и с матрицами ковариаций
и
соответственно:
где — объединенная ковариационная матрица,
— обратная объединенная ковариационная матрица,
и
— матрицы ковариаций двух классов,
и
— число точек в первом и во втором классах соответственно.
Причем в некоторой литературе также предлагается другой вариант:
Важно выбрать и применять только один из этих двух вариантов.
Кроме того, следует всегда отмечать, какой из вариантов используется.
Примечательно то, что при использовании второго варианта для первого класса с одной точкой и некоторого второго класса получается формула, аналогичная формуле расчета расстояния между точкой (не принадлежащей ни одному из классов) и классом (п. 2 «Расстояние Махаланобиса между двумя точками и между точкой и классом»):
Расстояние Евклида-Махаланобиса между двумя классами:
где — единичная матрица того же размера, что и
.
3.2 Алгоритм вычисления расстояния между двумя классами
Шаг 1. Вычислить математические ожидания значений признаков точек классов.
Шаг 2. Вычислить среднеквадратичные отклонения значений признаков точек классов.
Шаг 3. Вычислить ковариации между всеми парами признаков точек классов, составить ковариационные матрицы и объединенную ковариационную матрицу.
Шаг 4. Если матрица обратима, то вычислить расстояние по Махаланобису. Если матрица необратима, то вычислить расстояние по формуле расстояния Евклида — Махаланобиса.
3.3 Пример вычисления расстояния между двумя классами
Из примера п. 2.2.
Найдем расстояние между двумя классами.
Первые 3 аналогичных шага алгоритма были выполнены в п. 2.2.
Выполним 4 шаг.
Шаг 4. Если матрица обратима, то вычислим расстояние по Махаланобису. Если матрица необратима, то вычислим расстояние по формуле расстояния Евклида — Махаланобиса.
Объединенная ковариационная матрица:
Обратная объединенная ковариационная матрица:
• По принципу центроида
Расстояние Махаланобиса.
Расстояние Евклида — Махаланобиса .
Для варианта :
— расстояние Махаланобиса ;
— расстояние Евклида — Махаланобиса .
• По принципу ближнего соседа
Минимальное расстояние Махаланобиса между
и
и между
и
.
Минимальное расстояние Евклида — Махаланобиса между
и
и между
и
.
Для варианта :
— минимальное расстояние Махаланобиса между
и
и между
и
;
— минимальное расстояние Евклида — Махаланобиса между
и
и между
и
.
• По принципу дальнего соседа
Максимальное расстояние Махаланобиса между
и
и между
и
.
Максимальное расстояние Евклида — Махаланобиса между
и
и между
и
.
Для варианта :
— максимальное расстояние Махаланобиса между
и
и между
и
;
— максимальное расстояние Евклида — Махаланобиса между
и
и между
и
.
Код на Python 3.6 с использованием библиотеки numpy 1.19.5 для расчета представлен здесь.
4. Расстояние Махаланобиса и метод k-ближайших соседей
Классифицировать тестовую точку можно при помощи метода -ближайших соседей. Сначала нужно вычислить ковариационную матрицу для каждого класса, затем определить
-ближайших соседей для тестовой точки (вычислив расстояния от неё до всех точек всех классов с учетом ковариационных матриц) и отнести точку к классу с наибольшим количеством вхождений среди
ближайших соседей.
Далее представлен код программы для классификации методом -ближайших соседей.
Причем:
— Обратная матрица: (метрика Евклида — Махаланобиса);
— Используется квадрат расстояния (квадратный корень не имеет значения для конечного результата классификации, а без него программа работает немного быстрее).
Код на Python 3.6 с использованием библиотеки numpy 1.19.5
# Проверки убраны для удобочитаемости
import heapq
from collections import Counter
from operator import itemgetter
import numpy as np
class MkNN:
def __init__(self, k, classes, inv_cov_matrices):
self.n = len(classes)
self.k = k
self.classes = classes
self.inv_cov_matrices = inv_cov_matrices
@staticmethod
def mahalanobis_sqr(point_from, point_to, inverse_covariance_matrix):
delta = point_from - point_to
return max(np.float64(0), np.dot(np.dot(delta, inverse_covariance_matrix), delta))
def _get_k_smallest(self, test_point):
generator = (
(MkNN.mahalanobis_sqr(test_point, point, inv_cov), i)
for i, (class_, inv_cov) in enumerate(zip(self.classes, self.inv_cov_matrices))
for point in class_
)
return heapq.nsmallest(self.k, generator, key=itemgetter(0))
def predict(self, test_point):
return heapq.nlargest(1, Counter((i for _, i in self._get_k_smallest(test_point))).items(),
key=lambda t: (t[1], -t[0]))[0][0]
def predict_proba(self, test_point):
most_common = Counter((i for _, i in self._get_k_smallest(test_point)))
classes_proba = np.array([most_common.get(i, 0) for i in range(self.n)])
return classes_proba / classes_proba.sum()
def predict_all_max(self, test_point):
p = self.predict_proba(test_point)
return np.where(p == max(p))[0]
def main():
# Тестовые точки, которые следует отнести к какому-либо из classes
test_points = np.array([[4., 2.]])
# Классы с точками
classes = [
np.array([[1., 1.], [2., 2.], [3., 3.], [4., 4.], [5., 5.]]),
np.array([[3., 1.], [4., 0.], [6., 0.], [6., 2.], [5., 3.]])
]
# Число тренировочных точек
n_train_points = sum(class_.shape[0] for class_ in classes)
# Список матриц ковариаций для каждого класса
cov_matrices = [np.cov(class_, rowvar=False, ddof=1) for class_ in classes]
# Список обратных матриц ковариаций для каждого класса -- расстояние Евклида - Махаланобиса
inv_cov_matrices = [np.linalg.inv(cov + np.identity(cov.shape[0])) for cov in cov_matrices]
for test_point in test_points:
print("Point:", test_point)
# k от 1 до числа точек (не включая)
for i in range(1, n_train_points):
classifier = MkNN(i, classes, inv_cov_matrices)
print(f"{i}nn:",
1 + classifier.predict(test_point),
classifier.predict_proba(test_point),
classifier.predict_all_max(test_point))
if __name__ == "__main__":
main()
Вывод программы в формате:
"knn: [наименьший номер класса (с 1), к которому можно отнести точку] [вероятностные оценки для тестовых точек для всех классов] [индексы классов (с 0), к которым можно отнести точку]".
Point: [4. 2.]
1nn: 2 [0. 1.] [1]
2nn: 2 [0. 1.] [1]
3nn: 2 [0. 1.] [1]
4nn: 2 [0. 1.] [1]
5nn: 2 [0.2 0.8] [1]
6nn: 2 [0.33333333 0.66666667] [1]
7nn: 2 [0.42857143 0.57142857] [1]
8nn: 1 [0.5 0.5] [0 1]
9nn: 2 [0.44444444 0.55555556] [1]Визуализация классификации сетки точек при разных значениях представлена на рис. 4.

Код для визуализации на Python 3.6
# ...
from operator import sub
import numpy as np # 1.19.5
from matplotlib import colors, pyplot as plt # 3.3.4
# Визуализация классификации сетки точек
def show_data_on_mesh(k, classes, inv_cov_matrices):
# Генерация сетки
min_ = np.min([np.min(class_, axis=0) for class_ in classes], axis=1) - 1
max_ = np.max([np.max(class_, axis=0) for class_ in classes], axis=1) + 1
min_c = min(min_[0], min_[1])
max_c = max(max_[0], max_[1])
h = 0.05
test_mesh = np.meshgrid(np.arange(min_c, max_c, h), np.arange(min_c, max_c, h))
test_points = np.c_[test_mesh[0].ravel(), test_mesh[1].ravel()]
# Классификация точек сетки
classifier = MkNN(k, classes, inv_cov_matrices)
test_mesh_labels = [sub(*classifier.predict_proba(x)) for x in test_points]
# Создание графика
plt.figure(figsize=(6, 5), dpi=90)
class_colormap = colors.ListedColormap(['#070648', '#480607'])
plt.pcolormesh(test_mesh[0], test_mesh[1],
np.asarray(test_mesh_labels).reshape(test_mesh[0].shape),
cmap='coolwarm', shading='nearest')
plt.colorbar()
plt.scatter([point[0] for class_ in classes for point in class_],
[point[1] for class_ in classes for point in class_],
c=[-i for i, class_ in enumerate(classes) for _ in class_],
cmap=class_colormap)
plt.axis([min_c, max_c, min_c, max_c])
plt.xlabel("X")
plt.ylabel("Y")
plt.title("k=" + str(k))
plt.show()
# ...
Один запуск — один график для аргумента .
5. Взвешенное расстояние Махаланобиса
Расширением понятия расстояния Махаланобиса является взвешенное расстояние Махаланобиса, формула которой отличается от последней наличием дополнительного сомножителя — симметрической неотрицательно определенной матрицы весовых коэффициентов , недиагональные элементы которой чаще всего равны нулю:
Пример матрицы весовых коэффициентов (является единичной, поэтому на результат не повлияет) для точек с двумя признаками:
Формула взвешенного расстояния Евклида — Махаланобиса:
6. Заключение
В статье даны определения расстояний и метрик Махаланобиса, приведены теоретические сведения и рассмотрены примеры вычисления расстояний между двумя точками (также между точкой и классом) и между двумя классами, написана программа для классификации по методу -ближайших соседей с использованием метрики Евклида — Махаланобиса.
Что ещё?
1. Существует полиномиальное расстояние Махаланобиса, о котором можно почитать здесь.
2. О применении расстояния Махаланобиса в машинном обучении можно почитать в книге «Metric Learning» (авторы: Aurélien Bellet, Amaury Habrard, Marc Sebban; версия для ознакомления).
3. Расстояние Махаланобиса может использоваться в кластеризации (например, k-means: статья 1, статья 2, статья 3).
4. Расстояние Махаланобиса может использоваться в области интеллектуального анализа текста (статья).
5. Расстояние Махаланобиса может использоваться для обнаружения выбросов (статья 1, статья 2, статья 3).
Если есть замечания или ошибки, пишите на почту quwarm@gmail.com или в комментариях.