Компьютерное моделирование биомолекул уходит корнями в изучение принципов молекулярной динамики, которое имело место уже в 1950-х годах. В 1957 году впервые был опубликован компьютерный расчет молекулярной динамики, а спустя всего 20 лет первый расчет динамики белковой глобулы.
Известный физик Ричард Фейнман однажды отметил,
что все состоит из атомов, и что все, что происходит в живом объекте, можно представить в терминах колебаний и смещений атомов
Это значит, что для того, чтобы понять механизмы, которые лежат в основе функционирования живой материи, достаточно изучить динамическое поведение молекулярной системы. Или, что еще проще, достаточно свести решение биологической задачи к решению задачи молекулярной механики. Эта идея положила начало в развитии молекулярной динамики биомолекул.
Сегодня, одной из интересных задач компьютерного моделирования в биологии является изучение поведения биомолекул в различных растворах: динамика их структуры в ходе некоторого процесса и взаимодействие с другими молекулами. Эти задачи очень важны не только в фундаментальной науке, но и в фармацевтике. Например, перед проведением клинических испытаний разработанный препарат в первую очередь проходит этап молекулярного моделирования, на основе которого можно сделать вывод о том, как лекарство поведет себя в живом организме. К тому же, изучение молекулярной динамики может быть не только серьезным, но и интересным.
Когда-то компьютерная графика была малодоступным способом отображения молекул, однако сейчас все иначе. Одно дело произвести математический расчет положений и траекторий атомов в пространстве, другое же дело иметь возможность увидеть все своими глазами. Именно для этого существует ряд инструментов, которые позволяют биологам визуализировать расчеты молекулярной динамики. Однако, важно отметить, что визуализация - это лишь первый шаг на пути анализа результатов молекулярного моделирования.
Библиотека nglview
Одним из популярных и полезных инструментов для визуализации и анализа молекулярного моделирования является библиотека nglview, написанная на Python. Она представляет собой интерактивный виджет IPython/Jupyter, который позволяет визуализировать молекулярную структуру и траектории движения атомов.
Для отрисовки используется сервис NGL Viewer, который содержит в себе комплекс инструментов для молекулярной графики.
Также, важно отметить, что nglview позволяет визуализировать структуры как из файлов, RCSB PDB, simpletraj, так и из объектов, которые являются результатом анализа следующих библиотек: mdtraj, pytraj, mdanalysis, ParmEd, rdkit, ase, HTMD, biopython, cctbx, pyrosetta, schrodinger’s Structure.
Установка nglview
Установить nglview не сложно:
pip install nglview
jupyter-nbextension enable nglview --py --sys-prefixТакже пакет доступен и в conda-forge:
conda install nglview -c conda-forge
# might need: jupyter-nbextension enable nglview --py --sys-prefix
# if you already installed nglview, you can `upgrade`
conda upgrade nglview --force
# might need: jupyter-nbextension enable nglview --py --sys-prefixАх, да, документацию к nglview можно найти вот здесь. Тут же можно увидеть первый простейший пример кода для визуализации структуры 3pqr (кристаллическая структура метародопсина II в комплексе с С-концевым пептидом, полученным из Galpha-субъединицы трансдуцина) из RCSB PDB. Используя функцию show_pdbid() можно посмотреть на структуру, имея лишь идентификатор PDB:
import nglview as nv
view = nv.show_pdbid("3pqr") # load "3pqr" from RCSB PDB and display viewer widget
viewА вот и результат:

Метод demo()
Полезной функцией для новчиков может оказаться demo(), которая просто иллюстрирует готовый пример, а также позволяет оценить, что все установлено и настроено корректно для работы с библиотекой.
view = nv.demo()
view 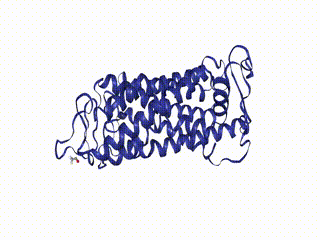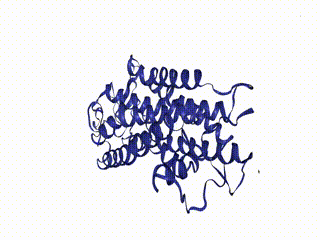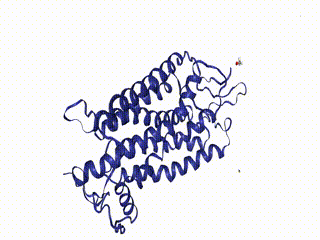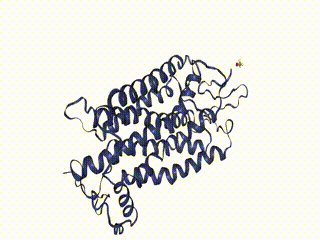
Это кристаллическая структура бычьего родопсина при разрешении 2,2 ангстрем. Доступная pdb-структура находится здесь. Тестовая структура лежит внутри библиотеки; путь к ней можно получить с помощью nv.datafiles.PDB, которая вернет /opt/miniconda3/envs/moldyn/lib/python3.7/site-packages/nglview/datafiles/md_1u19.pdb. Используя nv.datafiles можно получить и другие форматы тестового объекта: nv.datafiles.ALA3; nv.datafiles.ASE_Traj; nv.datafiles.GRO; nv.datafiles.MODULE_DIR; nv.datafiles.TRR; nv.datafiles.XTC.
Метод show_structure_file()
Чтобы посмотреть на структуру из тестового файла, достаточно применить функцию show_structure_file():
view = nv.show_structure_file(nv.datafiles.PDB)
viewЭтот код вернет нам тот же результат, что и выше.
Используя метод show_structure_file() можно также визуализировать из собственного файла. Для этого, в качестве примера, скачаем кристаллическую структуру человеческого B2-адренергического рецептора, связанного с G-белком, отсюда и посмотрим на нее с помощую nglview:
!wget https://files.rcsb.org/view/2rh1.pdb
view = nv.show_structure_file('2rh1.pdb')
viewРезультат:
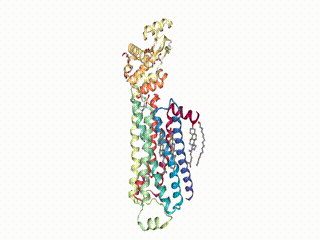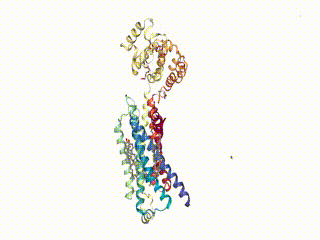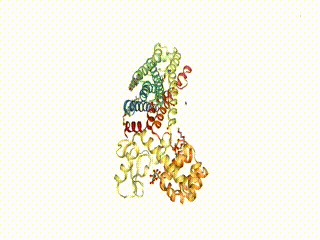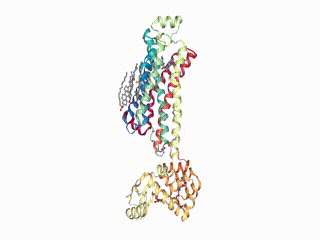
Методы add_cartoon() и remove_cartoon()
Все продемонстрированные ранее примеры содержат структуры в мультипликационном представлении, которое именуется cartoon. Давайте попробуем убрать из последней структуры все элементы, которое имею представление cartoon:
view.remove_cartoon()
А теперь, вернем их обратно:
view.add_cartoon()
Необходимо отметить, что из структуры на самом деле ничего не удаляется, мы всего лишь работаем с визуальным представлением.
Методы add_ball_and_stick() и remove_ball_and_stick()
Аналогично, методы add_ball_and_stick() и remove_ball_and_stick() позволяют удалять или добавлять элементы, имеющие шаро-стержневый, или ball_and_stick, вид:
view.remove_ball_and_stick()
Метод clear_representations()
Метод clear_representations() позволяет очистить виджет от отображения структуры. Результатом будет пустой виджет.
Изменение цвета и других параметров
В предыдущих примерах мы видели, что цвет поменялся, когда мы вновь добавили элементы в мультипликационном представлении. Так как в прошлый раз мы не указывали явно цветовую гамму, метод использовал параметр по умолчанию. Существует целый набор цветов для визуализации: atomindex, bfactor, chainid, chainindex, chainname, densityfit, electrostatic, element, entityindex, entitytype, geoquality, hydrophobicity, modelindex, moleculetype, occupancy, random, residueindex, resname, sstruc, uniform, value, volume.
Например, следующий код окрашивает молекулу на основе шкалы гидрофобности от красного к белому (Eisenberg et al., 1984):
view.add_cartoon(color='hydrophobicity')
В самом начале, в результате применения метода show_structure_file(), использовалась цветовая гамма atomindex. Получим теперь ее самостоятельно:
view.clear_representations()
view.add_cartoon(color='atomindex')
Также мы можем изменить прозрачность изображения. Для этого можно воспользоваться функцией update_cartoon():
view.update_cartoon(opacity=.6)
Выделение интересующего объекта
Чтобы выделить интересующие нас объекты, достаточно использовать "игру" с различными вариантами представления структур. Например, нас может заинтересовать конкретный лиганд CAU. Тогда можно воспользоваться тем, чтобы выделить его с помощью
представления, отличного от представления белка, напрнимер, вот так:
view.clear_representations()
view.add_cartoon(color='atomindex')
view.add_ball_and_stick('CAU')
view.remove_label()
view.add_label('CAU', radius=1.5, color='black', label_type='atomname')

По порядку. Сначала мы почистили виджет с помощью clear_representations(). Затем добавили рецептор с помощью add_cartoon() в цветовой гамме atomindex. Используя add_ball_and_stick() добавили лиганд CAU. Удалили (на всякий) все отметки с помощью remove_label() и добавили только те, которые нам интересны, в данном случае были добавлены названия атомов черного цвета в размере 1.5, с помощью add_label().
Метод _display_repr()
И в заключение, отмечу полезность такого метода как _display_repr(), который предоставляет меню ручного управления некоторыми параметрами виджета.
view._display_repr()
На самом деле о возможностях nglview рассказывать еще очень много, но, я думаю, что в рамках одной статьи этого будет достаточно. Пишите, если вас что-то заинтересовало, мне всегда будет приятно поделиться своими знаниями :)
