 Привет, Хабр! В этой части мы опишем вам алгоритм, с помощью которого были получены цвета на графах из первой части. В основе алгоритма лежит k-medoids — довольно простой и прозрачный метод. Он представляет собой вариант популярного k-means, про который наверняка большинство из вас уже имеет представление.
Привет, Хабр! В этой части мы опишем вам алгоритм, с помощью которого были получены цвета на графах из первой части. В основе алгоритма лежит k-medoids — довольно простой и прозрачный метод. Он представляет собой вариант популярного k-means, про который наверняка большинство из вас уже имеет представление. В отличие от k-means, в k-medoids в качестве центроидов может выступать не любая точка, а только какие-то из имеющихся наблюдений. Так как в графе между вершинами расстояние определить можно, k-medoids годится для кластеризации графа. Главная проблема этого метода — необходимость явного задания числа кластеров, то есть это не выделение сообществ (сommunity detection), а оптимальное разбиение на заданное количество частей (graph partitioning).
С этим можно бороться двумя путями:
- либо задавая некую метрику «качества» разбиения и автоматизируя процесс выбора оптимального числа кластеров;
- либо рисуя раскрашенный граф и пытаясь определить наиболее логичное разбиение «на глаз».
Второй вариант годится только для небольших данных и не более чем для пары десятков кластеров (или надо использовать алгоритм с многоуровневой кластеризацией). Чем крупнее граф, тем более грубыми деталями придётся довольствоваться при оценке качества «на глаз». При этом для каждого конкретного графа придётся повторять процедуру заново.
PAM
Самый распространённый вариант реализации k-medoids называется PAM (Partitioning Around Medoids). Он представляет собой «жадный» алгоритм с очень неэффективной эвристикой. Вот как он выглядит приложении к графу:
Вход: граф G, заданное число кластеров k
Выход: оптимальное разбиение на k кластеров + "центральная" вершина в каждом из них
Инициализируем: выбираем k случайных узлов в качестве медоидов.
Для каждой точки находим ближайший медоид, формируя исходное разбиение на кластеры.
minCost = функция потерь от исходной конфигурации
Пока медоиды не стабилизируются:
Для каждого медоида m:
Для каждой вершины v != m внутри кластера с центром в m:
Перемещаем медоид в v
Перераспределяем все вершины между новыми медоидами
cost = функция потерь по всему графу
Если cost < minCost:
Запоминаем медоиды
minCost = cost
Кладем медоид на место (в m)
Делаем наилучшую замену из всех найденных (т. е. внутри одного кластера меняем один медоид) -- это одна итерация.За каждую итерацию алгоритм перебирает
Самая жадная эвристика
Поработав немного с этим алгоритмом, мы стали думать, как ускорить процесс, ибо кластеризовать граф из всего 1200 узлов на одной машине занимало несколько минут времени (для 10000 узлов — уже несколько часов). Одно из возможных улучшений состоит в том, чтобы сделать алгоритм еще более жадным, проводя поиск наилучшей замены только по одному кластеру, а ещё лучше — по маленькой доле точек внутри одного кластера. Последний вариант эвристики придает алгоритму следующий вид:
Вход: граф G, заданное число кластеров k
Выход: оптимальное разбиение на k кластеров + "центральная" вершина в каждом из них
Для каждой точки находим ближайший медоид, формируя исходное разбиение на кластеры.
Сколько итераций подряд не было улучшения: StableSequence = 0
While True:
Для каждого медоида m:
Случайно выбираем s точек внутри кластера с центром в m
Для каждой вершины v из s:
Перемещаем медоид в v
Перераспределяем все вершины между новыми медоидами
cost = функция потерь по всему графу
Если cost < minCost:
Запоминаем медоиды
minCost = cost
Кладем медоид на место (в m)
Если наилучшая замена из s улучшает функцию потерь:
Производим эту замену
StableSequence = 0
Иначе:
StableSequence += 1
Если StableSequence > порога:
Возвращаем текущую конфигурациюПростым языком, теперь мы ищем оптимальную замену не по всем вершинам графа, а по одному кластеру, причем перебираем не все вершины внутри этого кластера, а лишь
Оказывается, его снижение до 2 и даже до 1 практически не ухудшает различные метрики качества кластеров (например, модулярность). Поэтому мы решили взять
Clara
Следующий шаг к улучшению масштабируемости k-medoids — это довольно известная модификация PAM, называемая clara. Из исходного графа случайным образом выбирается подмножество вершин, и кластеризуется подграф, образованный этими вершинами. Затем (в предположении связности графа) оставшиеся вершины просто распределяются по ближайшим медоидам из подграфа. Вся соль clara состоит в последовательном прогоне алгоритма на разных подмножествах вершин и выборе наиболее оптимального из результатов. За счет этого предполагается компенсировать ущерб от исключения части информации при каждом отдельном прогоне, а также избежать застревания в локальном минимуме. В качестве меры оптимальности при выборе результата мы использовали модулярность. Можно придумать много хитрых способов вычленения подграфа на первом этапе, но мы использовали несколько простейших:
- Случайным выбором вершин с равной вероятностью;
- С вероятностью, пропорциональной степени вершины в исходном графе;
- Всегда выбирать фиксированное количество вершин с наибольшей степенью, а недостающие вершины — случайно с равной вероятностью;
- Выбирать пары вершин с мерой Жаккарда между ними выше порога, а недостающее с равной вероятностью (либо ползти по соседям).
Все способы показали приблизительно одинаковое качество кластеров по популярной метрике WTF, которая равна числу возгласов «What the fuck?» при визуальном просмотре состава кластеров (например, при попадании в один кластер форума ВДВ и сайта cosmo.ru).
Выбор объёма подвыборки для clara также представляет собой компромисс между качеством и скоростью. Чем больше кластеров присутствует в данных, тем меньше наши возможности для семплинга: некоторые кластеры могут просто не попасть в выборку. Если разница в размерах кластеров большая, то такой подход вообще противопоказан. Если же структура более-менее равномерная, то мы рекомендуем семплировать
По всей видимости, clara изначально предназначался для уменьшения времени выполнения алгоритмов кластеризации, чья вычислительная сложность растет быстрее, чем число вершин (когда прогнать несколько раз на подграфе быстрее, чем один раз на полном графе). Поэтому польза clara при использовании улучшенной эвристики (линейная сложность) резко падает. Однако мы нашли другое применение clara: ансамблевый подход, о котором речь пойдет позже.
Локальное прореживание (L-Spar)
Следующий шаг называется L-Spar (от local sparsification), и он подробно описан здесь. Это метод предобработки графа перед кластеризацией. Он «прореживает» граф путем удаления части ребер (но не узлов!), не разрушая, как правило, его связность.
Чтобы реализовать этот шаг, нужно знать степень «схожести» между двумя любыми узлами. Так как нам потребуется схожесть на каждой итерации k-medoids, мы решили считать заранее матрицу схожести и сохранять ее на диск. В качестве меры схожести была использована мера Жаккарда, с которой вы, скорее всего, уже встречались:
где
Алгоритм L-Spar очень прост. Сначала для каждого узла
Доказано, что при этом сохраняется степенной закон распределения степеней в графе, приводящий к феномену «тесного мира» или «small world», когда кратчайший путь между двумя узлами в среднем имеет очень маленькую длину. Это свойство присуще большинству сетей, порожденных человеком, и L-Spar не рушит его.
L-Spar обладает важным преимуществом перед, например, обрубанием всех ребер с
Данный метод предобработки графа, естественно, можно использовать при любом методе кластеризации. Самый большой эффект может быть достигнут для алгоритмов, чья сложность зависит только от числа ребер, но не узлов, так как сокращается только число ребер. В этом плане k-medoids не повезло: его сложность зависит только от числа узлов, однако при более отчетливой структуре сообществ может уменьшиться число итераций, необходимых для сходимости. Если sparsification удаляет «шумные» ребра, то можно ожидать более отчетливой структуры сообществ, а значит, и уменьшения числа итераций. Эту гипотезу подтвердили эксперименты авторов этого метода, но не подтвердили наши эксперименты.
В нашем графе доменов «шумных» рёбер нет, так как слабые аффинити между вершинами были заранее отфильтрованы. Если кластеры плохо выделяются визуально, они плохо выделяются и в данных. Поэтому для наших графов число итераций k-medoids не зависит от степени прорежения. При
Стоит отметить еще одно свойство L-Spar, которое мы использовали вовсю: улучшение читабельности картинки. Из-за радикального снижения числа ребер теперь, возможно, удастся лучше визуализировать Волосяные Шары социальных сетей и показывать сообщества на них. Пример такой визуализации был приведен в предыдущей части данного поста, где был граф из 1285 веб-доменов после применения L-Spar и до него.
Выбор числа кластеров 
Одна из проблем k-medoids — фиксированное количество кластеров. Для нахождения оптимального числа кластеров мы пробовали несколько вариантов. Беда с модулярностью, проводимостью, normalized cut и другими метриками в том, что они страдают от лимита разрешения, и их максимум достигается на слишком низких
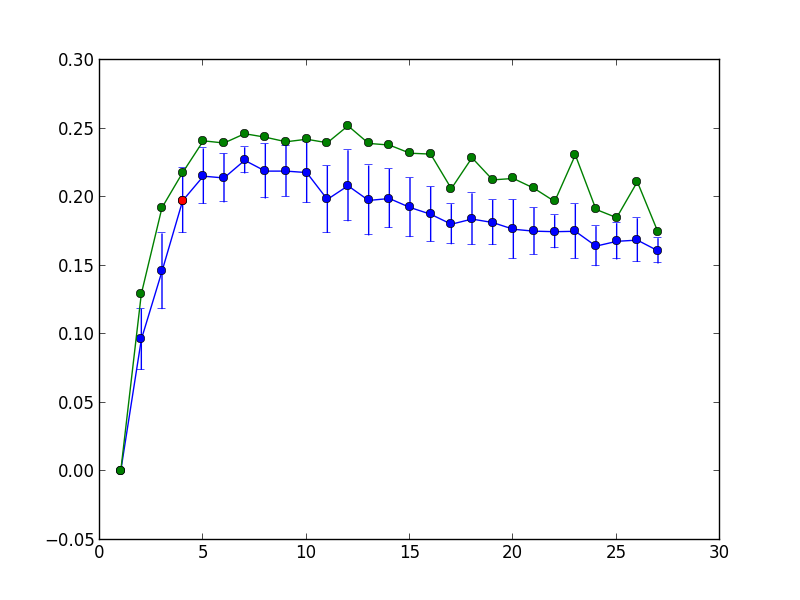
Синим цветом показана средняя модулярность и её 1.95 стандартных отклонений на 100 повторениях k-medoids, зелёным — максимальное значение модулярности на 100 повторениях. Красным цветом выделена точка, соответствующая максимальному количеству кластеров
Стабильные ядра
Вышеописанные модификации могут быть хороши в плане масштабируемости, однако они приводят к двум интересным проблемам. Во-первых, может упасть качество кластеров: неочевидно, насколько падает логичность разбиения при упрощении эвристики (тем более, при выделении подграфа или удалении большей части ребер). Во-вторых, под угрозой стабильность результата: разбиение на кластеры при втором прогоне будет существенно отличаться от разбиения на первом, даже при одних и тех же исходных данных. Все наши модификации еще сильнее рандомизировали алгоритм. А что если граф вдобавок эволюционирует во времени? Как отождествлять друг с другом кластеры из разных прогонов?
Мы провели небольшой эксперимент с графом доменов из 1209 узлов, чтобы проверить стабильность k-medoids. Для двух разных прогонов алгоритма со всеми модификациями мы нашли наиболее «центральные» узлы (по метрике harmonic centrality) внутри каждого кластера — четверть от общего числа узлов в кластере, но не меньше восьми. Затем для каждого кластера из первого разбиения мы нашли кластер второго разбиения, куда входит наибольшее число его «центральных» узлов. Если
Данной проблеме посвящены статьи: один, два, три. В них говорится о том, что добавление или удаление всего одной вершины может радикально поменять все разбиение, влияя не только на ближайших соседей, но и на отдаленные части графа. Одно из решений этой проблемы — усреднение кластеризаций. Это попытка адаптировать ансамблевый подход, хорошо зарекомендовавший себя в обучении с учителем, для задач кластеризации. Если сделать не один, а, скажем, 100 прогонов алгоритма, а затем найти группы вершин (ядра), которые всегда попадали вместе в один и тот же кластер, то эти группы будут гораздо стабильнее во времени, чем результаты индивидуальных прогонов. Единственный недостаток этого метода — скорость: десятки повторений могут свести на нет достижения по убыстрению алгоритма. Однако при нахождении ядер можно семплировать граф с меньшими потерями, т.е. использовать clara.
Мы реализовали немного иной вариант получения ядер. Для каждой пары вершин мы считаем, в какой доле прогонов они попадали в один и тот же кластер. Полученные данные могут быть интерпретированы как вес ребер (степень близости) в новом, полном графе. Далее по этому графу строится иерархическая кластеризация. Мы использовали агломеративный метод scipy.linkage и метод Ward в качестве функции расстояния при объединении кластеров.
Здесь можно посмотреть на полную версию дендрограммы.
Порог отсечения можно установить на любой высоте, получая при этом разное число кластеров. Может показаться наиболее логичным ставить такой порог, чтобы число кластеров было равно числу кластеров в каждом прогоне k-medoids при получении самой дендрограммы. Однако на практике, как ни странно, если для k-medoids подобрать верное
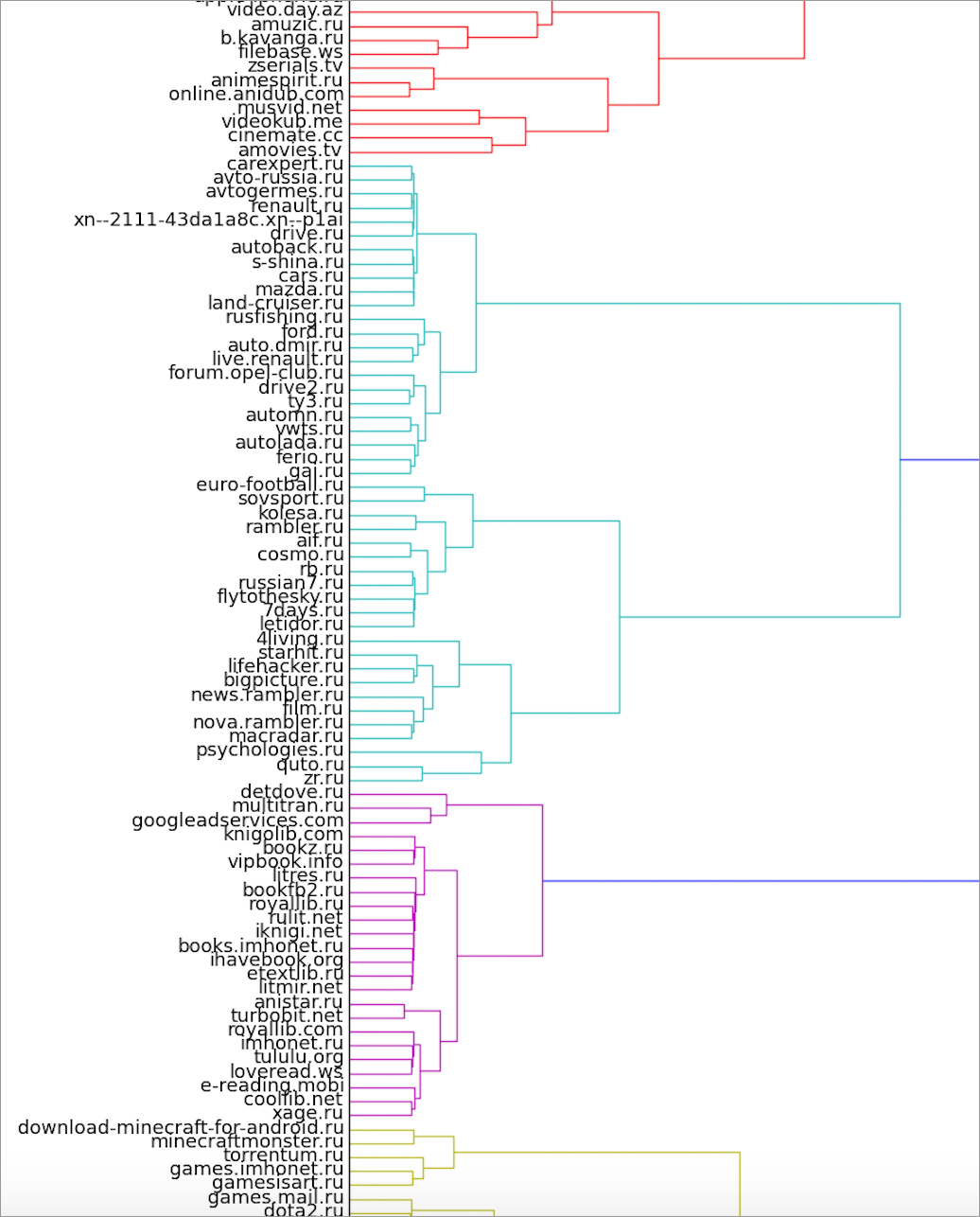
В кадр попали также кластер книг и кусочки игрового и сериального кластеров
Ядра доменов, полученные данным способом, успешно функционируют как сегменты пользователей в нашей RTB-системе Exebid.DCA.
Уже существует несколько отличных алгоритмов выделения сообществ, быстрее и эффективнее, чем наши наработки. Louvain, MLR-MCL, SCD, Infomap, Spinner, Stochastic Blockmodeling — некоторые из них. В будущем планируем написать о наших экспериментах с этими методами. Но нам хотелось написать что-то самим. Вот некоторые из направлений улучшения:
- Вычисление меры Жаккарда — самый ресурсоемкий этап, требующий
операций. Известен быстрый приближенный метод получения схожестей под названием MinHash. Почитать про него можно, например, здесь. Еще есть более сложные методы, например, BayesLSH.
- Вычислительная сложность, зависящая от числа кластеров, — эта особенность k-medoids делает его неприменимым для большого спектра задач. С ней можно бороться так: для каждого медоида на каждой итерации находить и сохранять список «соседних» медоидов, и только их пересчитывать при каждой итерации.
- Произвол выбора количества кластеров
— еще одна сложность с k-medoids. Один из выходов — найти стоящую метрику, по которой сверять результаты для разных
. На эту роль претендует WCC (Weighted Community Clustering coefficient), основанный на анализе замкнутых треугольников внутри и вне кластеров. Возможно, на эту метрику стоит перевести и выбор оптимальной замены на каждой итерации.
Спасибо за внимание!
Только зарегистрированные пользователи могут участвовать в опросе. Войдите, пожалуйста.
Чему посвятить следующие статьи цикла?
77.94% Подробному разбору новейших алгоритмов кластеризации53
51.47% Экспериментам с другими алгоритмами, с примерами и кодом35
11.76% Новым усовершенствованиям k-medoids8
20.59% Туториалу по визуализации в networkx14
26.47% Туториалу по визуализации в graph-tool или gephi18
2.94% Ничему. Рыбачук — это печально.2
Проголосовали 68 пользователей. Воздержались 23 пользователя.