Пересечение медицины и науки о данных всегда было актуальным; возможно, самый очевидный пример — реализация нейронных сетей в глубоком обучении. По мере развития науки о данных и машинного обучения будет развиваться и медицина, но верно и обратное.
Нанотехнологии, стволовые клетки, оптогенетика, метаболомика, редактирование генов и интерфейсы мозг — компьютер — вот лишь некоторые области, выигрывающие от взаимовыгодных отношений медицины и науки о данных, представители которых должны научиться расти и адаптироваться к эволюции в своей сфере — иначе они рискуют остаться позади. К старту курса по Machine Learning и Deep Learning делимся статьёй о возможностях пакета MNE для визуализации данных о мозге. По словам автора — нейрохирурга и спикера TEDx — как только MNE будет сопряжён с TensorFlow, sklearn или другой библиотекой машинного обучения, в интерфейсы мозг — компьютер сможет погрузиться любой человек.
Мозг интригует почти каждого, кто его рассматривает; он одновременно двусмыслен в своей запутанности и конкретен — в действии. Кроме того, сложность мозга требует изобретательности и творческого подхода при его изучении.
В своей предыдущей статье "Введение в интерфейсы мозг — компьютер"¹ я подробно описал некоторые сложности мозга и связанные с ними нейротехнологии. В этих сложностях бывает трудно разобраться, но определённые инструменты позволяют практически любому человеку работать с данными нейровизуализации и воспроизводить научные эксперименты².
Модуль MNE-Python³ — это пакет с открытым исходным кодом для просмотра данных от нейрофизиологических инструментов, один из немногих доступных инструментов, позволяющий просматривать, манипулировать и анализировать данные образцов ЭЭГ, ЭКоГ, МЭГ и других методов получения данных в режиме онлайн.
Эта библиотека очень полезна для визуализации многих этапов, используемых в различных системах интерфейсов мозг — компьютер, и для лучшего понимания этой развивающейся технологии. Рассмотрим возможности MNE и работу с наборами данных для тестирования этих возможностей.
MNE
1. Импорт модулей
Люди с опытом больше наверняка знакомы с Jupyter Labs и Anaconda, с которыми и следует работать при использовании MNE. Загрузка Anaconda и последующая установка MNE позволят вам использовать Jupyter Labs на localhost и значительно упростят программирование. При выполнении программы в Jupyter Labs вы можете разбить свой код на блоки, которые можно выполнять независимо друг от друга; это гораздо удобнее подхода типичных IDE.
Однако, как только программа будет готова, вы можете перенести её в IDE по выбору (убедитесь, что установили MNE в виртуальном окружении, если вы используете его) и запустить программу. Когда я переносил программу, то столкнулся с несколькими проблемами, в основном они решались загрузкой и импортом нескольких модулей.
import matplotlib
import matplotlib.pyplot as plt
import PyQt5
import pathlib
import mne
import mne_bids
from mne.datasets import sample
import picard
from surfer import Brain
import sobol_seq
import os.path
import os.path as op
from os import path
from mne.forward import read_forward_solution
from mne.minimum_norm import (make_inverse_operator, apply_inverse,
write_inverse_operator)
import nibabel
matplotlib.use('Qt5Agg')Я не был знаком с picard, surfer или sobol_seq, но они были необходимы для построения некоторых используемых графиков.
Седьмая строка: from mne.datasets import sample, собирает фундаментальный набор данных, используемый для изучения MNE. В этом наборе данных уже есть информация, чтобы любой человек мог начать работать с MNE. К сожалению, некоторые задачи, поставленные перед людьми, которые отдали свои данные для набора, были простыми, но в MNE есть множество встроенных наборов данных⁴.
Модуль mne_bids⁵ не использовался в моём коде, но это важная часть MNE, которая задействована в большей части кода.
Поскольку он очень полезен, я, конечно, решил не использовать его. Это упростит жизнь…
Этот модуль совместим со структурой данных визуализации мозга (BIDS)⁶, то есть с типичным способом организации нейрофизиологических данных. В частности, он определяет следующее:
Форматы файлов.
Как присваивать имена файлам.
Где размещать файлы в каталоге.
Какие дополнительные метаданные следует хранить.
выглядит это так:
out_path = pathlib.Path('out_data/sample_BIDS')
bids_path = mne_bids.BIDSPath(subject='01',
session='01',
task='audiovisual',
run='01',
root=out_path)
mne_bids.write_raw_bids(raw, bids_path=bids_path, events_data=events, event_id=event_id, overwrite=True)Папка out_data уже существует, в ней создаётся sample_BIDS с необходимой информацией о пациенте. События и event_id мы определили ранее.
2. Импорт необработанных данных
После того как мы импортировали данные нашей выборки, можно импортировать исходную информацию, которая будет необходима для дальнейшего анализа.
"""Creating data path"""
sample_data_dir = sample.data_path()
sample_data_dir = pathlib.Path(sample_data_dir)
"""Importing raw data"""
raw_path = sample_data_dir / 'MEG' / 'sample' / 'sample_audvis_raw.fif'
raw = mne.io.read_raw(raw_path)
print(raw.info['bads'])
"""Viewing raw data"""
raw.plot()
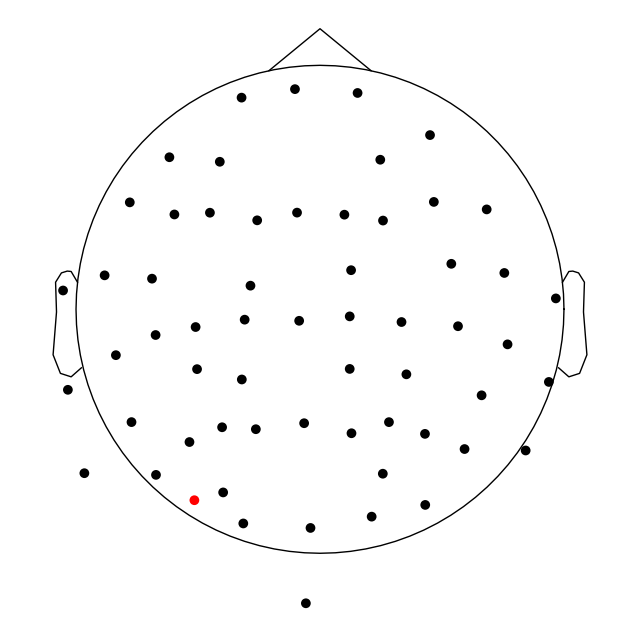
raw.plot_sensors(ch_type='eeg')
raw.plot_sensors(ch_type='eeg', kind='3d')
"""Finding events/event ids"""
events = mne.find_events(raw)
event_id = {
'Auditory/Left': 1,
'Auditory/Right': 2,
'Visual/Left': 3,
'Visual/Right': 4,
'Smiley': 5,
'Button': 32
}
total_auditory_events = len(events[events[:,2] == 1]) + len(events[events[:,2] == 2])
total_visual_events = len(events[events[:,2] == 3]) + len(events[events[:,2] == 4])
print(total_auditory_events, total_visual_events)После создания пути данных и навигации по данным мы можем построить их различными способами.
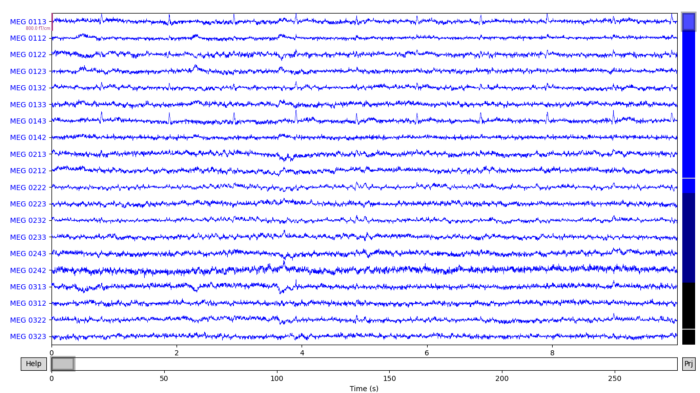
Создаваемый график интерактивен, это сильно облегчает жизнь. Например, электрод ЭЭГ 053 выглядит как выброс, который я не хочу видеть в составе своих данных. Я могу просто щёлкнуть канал, щелчок пометит его как "плохой". Если вы хотите увеличить масштаб данных, достаточно использовать функциональную клавишу и клавиши со стрелками (по крайней мере на mac).
Я также могу нанести электроды на модель головы. Следует отметить, что изображение, созданное с помощью Jupyter Labs и PyCharm, было другим. PyCharm применялся для создания изображения ниже, но некоторые датчики, кажется, немного смещены.
Ничего страшного, если датчики не находятся непосредственно на голове — это означает, что они могут быть на ушах, шее или где-то ещё, однако это необходимо учитывать при построении графиков.
Также можно создать 3D-модель.

Нижняя часть приведённого выше кода была посвящена поиску различных событий на основе каналов стимулов. Событиям, которые были обнаружены MNE, присвоены номера 1, 2, 3, 5 и 32. Эти номера связаны с конкретным стимулом — благодаря документации MNE были найдены и обозначены правильные стимулы. Я использовал эту информацию, чтобы создать график сырых данных с аннотациями при помощи датчиков ЭЭГ.
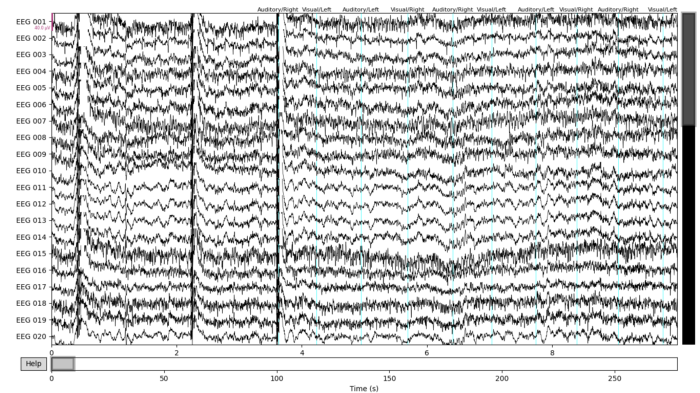
Общее количество слуховых событий составило 145, а общее количество зрительных событий — 144.
3. Фильтрация необработанных данных и построение спектральной плотности мощности
Прежде чем двигаться дальше, отфильтруем данные, чтобы они выглядели намного чище и их проще было анализировать.
"""Applying filters and plotting PSDs"""
raw_eeg = raw.copy().pick_types(meg=False, eog=False, eeg=True)
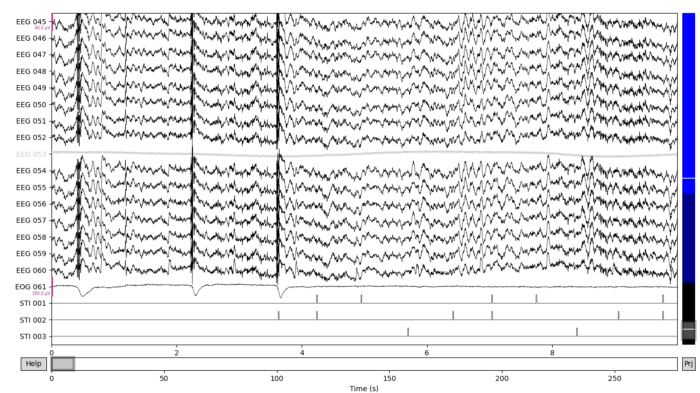
raw_eeg.plot(events=events, event_id=event_id, title="Unfiltered EEG")
raw_eeg.load_data()
raw_eeg_filtered = raw_eeg.copy().filter(l_freq=0.1, h_freq=40)
raw_eeg_filtered.plot(events=events, event_id=event_id, title="Filtered EEG")
fig, ax = plt.subplots(2)
raw_eeg.plot_psd(ax=ax[0], show=False)
raw_eeg_filtered.plot_psd(ax=ax[1], show=False)
ax[0].set_title('PSD before filtering')
ax[1].set_title('PSD after filtering')
ax[1].set_xlabel('Frequency (Hz)')
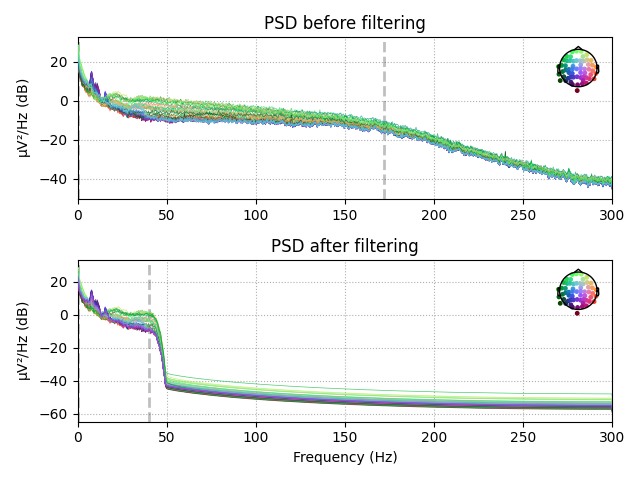
fig.set_tight_layout(True)Я использовал фильтр низких и высоких частот с частотами 40 Гц и 0,1 Гц. Частоты ниже 40 Гц и выше 0,1 Гц сохранены, другие — удалены. Используемые частоты будут различаться в зависимости от эксперимента, но эти значения для данного набора — это хорошие значения по умолчанию.

После получения нефильтрованных и фильтрованных данных я могу построить график спектральной плотности мощности сигнала (далее — PSD), полученного от разных электродов. PSD — это мощность сигнала как функция частоты, по частотам
4. Эпохи и вызванная информация
Как уже упоминалось в предыдущей статье, эпохи — это просто сегментация данных на несколько различных частей. Вызванные объекты — это усреднённые сигналы нескольких эпох ЭЭГ или МЭГ, такая стратегия обычна при оценке вызванной стимулом активности.
tmin = -0.250
tmax = 0.8
baseline = (-0.2, 0)
epochs = mne.Epochs(raw_eeg_filtered,
events=events,
event_id=event_id,
tmin=tmin,
tmax=tmax,
baseline=baseline,
preload=True)
print("\nEpochs Info:")
print(epochs)
epochs.save(pathlib.Path('out_data') / 'epochs_epo.fif',
overwrite=True)
epochs['Visual'].plot()
"""Evoked Data Information"""
evoked_auditory = epochs['Auditory'].average()
evoked_visual = epochs['Visual'].average()
evoked_visual.plot(spatial_colors=True)
evoked_visual.plot_topomap(ch_type='eeg')
mne.viz.plot_compare_evokeds([evoked_auditory, evoked_visual], picks='eeg')
mne.write_evokeds(fname=pathlib.Path('out_data') / 'evokeds_ave.fif',
evoked=[evoked_auditory, evoked_visual])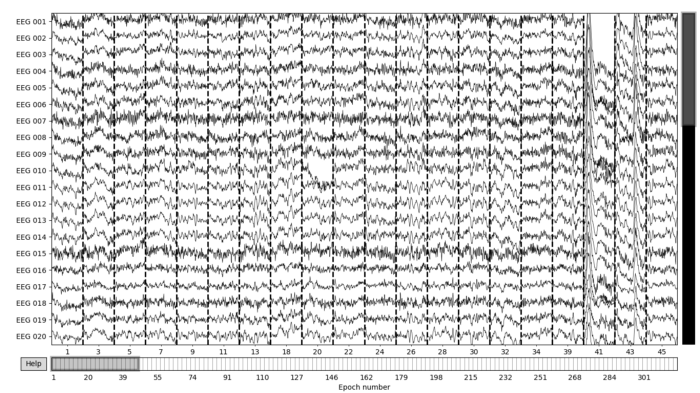
Хотя я продолжаю использовать данные ЭЭГ, в этом нет необходимости. Можно также использовать исходные необработанные данные с информацией о МЭГ и разделить данные на эпохи. В дальнейшем их можно очистить с помощью независимого компонентного анализа (ICA). Чтобы использовать исходные необработанные данные, вместо raw_eeg_filtered я задействую raw.

Я также могу построить график вызванных данных относительно каждого метода получения данных.
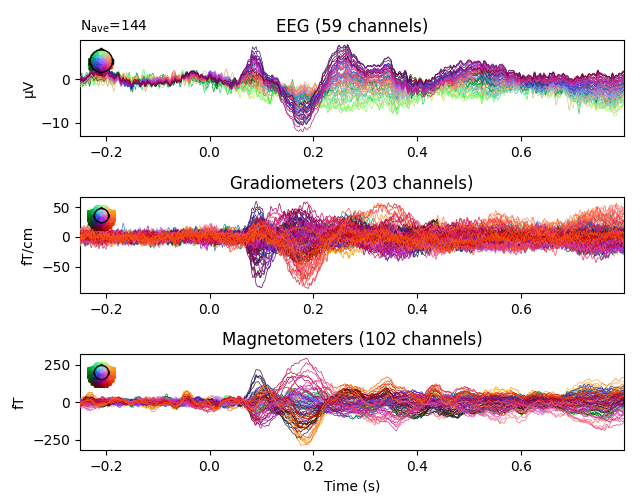
Топокарта также может быть создана с использованием вызванных данных для просмотра напряжения на различных участках головы от начала до конца эпохи.
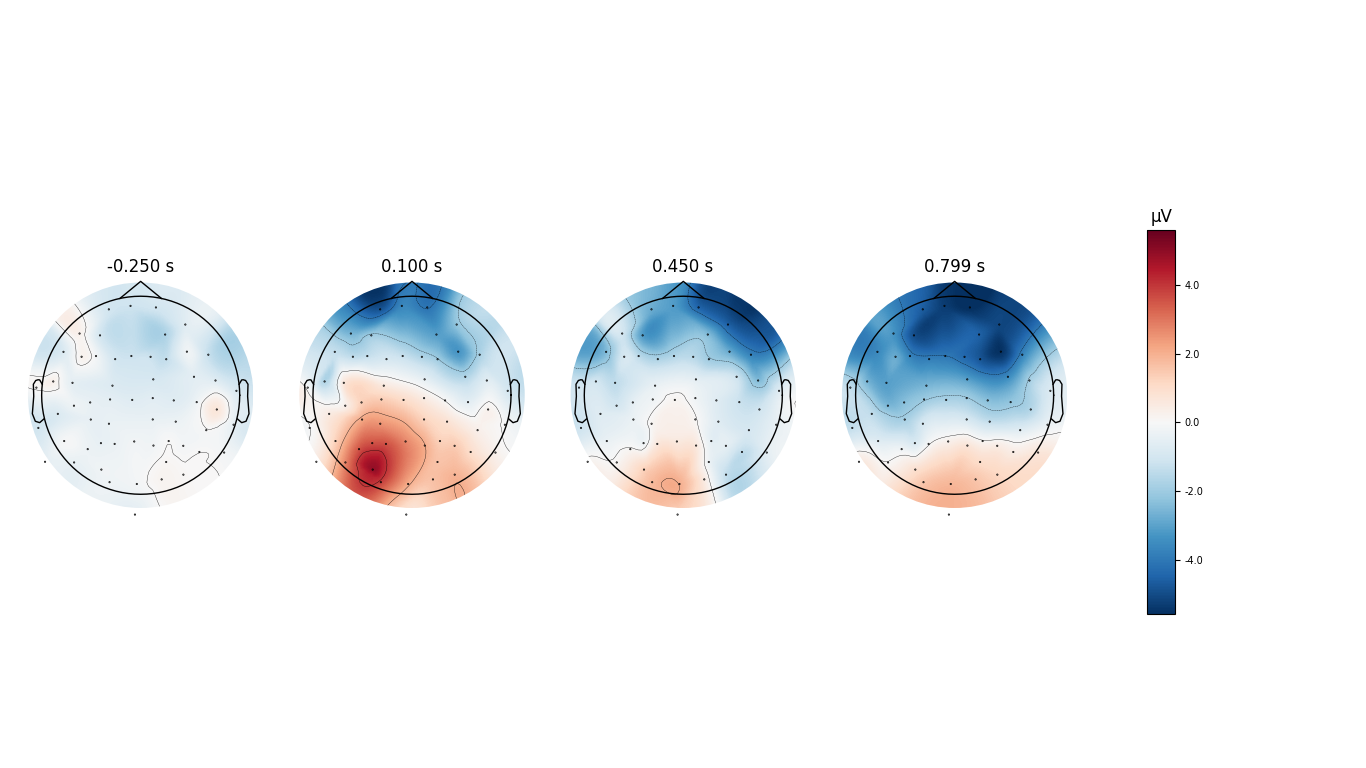
Я также могу сравнить вызванную информацию связанных с различными стимулами эпох.

4. Применение независимого компонентного анализа (ICA)
Как объяснялось в моей предыдущей статье, ICA — это метод, используемый для очистки эпох, не входящий в состав основных методов фильтрации, применяемых к исходным данным. С помощью ICA можно найти и устранить определённые артефакты, включая улавливаемые электродами компоненты ЭОГ и ЭКГ.
"""Cleaning Epochs with Independent Component Analysis (ICA) """
if path.exists('out_data/epochs_cleaned.fif') == False:
tmin = epochs.tmin
tmax = epochs.tmax
baseline = (None, 0)
epochs_ica = mne.Epochs(raw,
events=events,
event_id=event_id,
tmin=tmin,
tmax=tmax,
baseline=baseline,
preload=True)
print("\nEpochs ICA Info: ")
print(epochs_ica)
n_components = 0.8 # Should normally be higher, like 0.999!!
method = 'picard'
max_iter = 100 # Should normally be higher, like 500 or even 1000!!
fit_params = dict(fastica_it=5)
random_state = 42
ica = mne.preprocessing.ICA(n_components=n_components,method=method,max_iter=max_iter,fit_params=fit_params,random_state=random_state)
ica.fit(epochs_ica)
ica.plot_components(inst=epochs)
ecg_epochs = mne.preprocessing.create_ecg_epochs(raw, reject=None,
baseline=(None, -0.2),
tmin=-0.5, tmax=0.5)
ecg_evoked = ecg_epochs.average()
ecg_inds, ecg_scores = ica.find_bads_ecg(
ecg_epochs, method='ctps')
eog_epochs = mne.preprocessing.create_eog_epochs(raw, reject=None,
baseline=(None, -0.2),
tmin=-0.5, tmax=0.5)
eog_evoked = eog_epochs.average()
eog_inds, eog_scores = ica.find_bads_eog(
eog_epochs)
components_to_exclude = ecg_inds + eog_inds
ica.exclude = components_to_exclude
ica.plot_scores(ecg_scores)
ica.plot_sources(ecg_evoked)
ica.plot_overlay(ecg_evoked)
# Running ICA on previous data saved
epochs_cleaned = ica.apply(epochs.copy())
epochs_cleaned.plot(title="After ICA")
epochs.plot(title="Before ICA")
epochs_cleaned.save(pathlib.Path('out_data') / 'epochs_cleaned.fif',
overwrite=True)
epochs_visual = epochs_cleaned["Visual"]Оператор if нужен только для того, чтобы эта часть кода не запускалась каждый раз в IDE. Это излишне в Jupyter Labs, но удобно после того, как очищенные эпохи уже сохранены: значительно сокращается время выполнения.

После применения ICA мы можем просмотреть эпохи до и после.

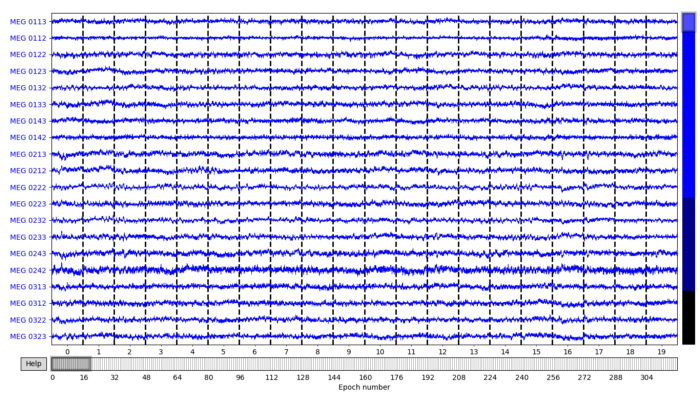
Мы также можем просмотреть оценки компонентов ICA. Это позволяет определить, какие компоненты с наибольшей вероятностью связаны с различными артефактами.

Одним из компонентов, который показал высокую вероятность связи с артефактом, был компонент 001, его можно проанализировать.

Мы также можем просмотреть эпохи до и после очистки другим способом, то есть просмотреть сигнал для каждого метода сбора до и после применения ICA.
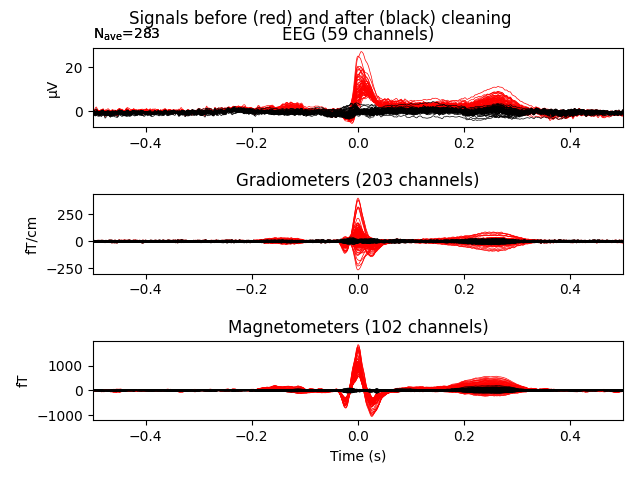
5. Временно-частотный анализ
Временно-частотный анализ также возможно сделать через MNE, но я использую эту функцию не так часто. Однако особенно интересная часть этого этапа для меня — просмотр наличия различных мозговых волн на разных частях черепа. На основе найденной частоты можно определить различные мозговые волны.
код
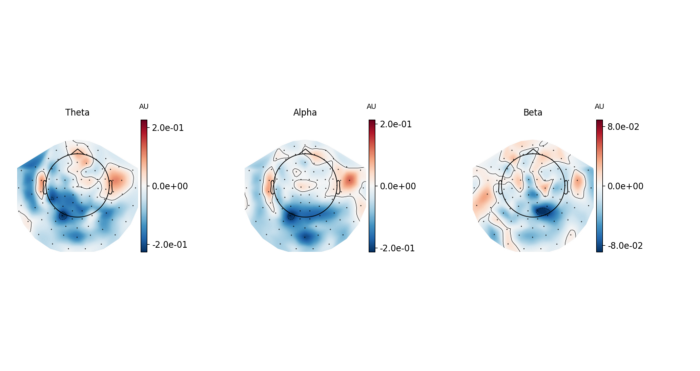
6. Визуализация данных сканов МРТ и датчиков ЭЭГ
Один из самых интересных моментов в MNE заключается в том, что источник сигнала можно отследить, выполнив несколько шагов. Эти шаги включают следующее:
Энергетический метаболизм мозга.
Трансинформация.
Расчёт прямого решения.
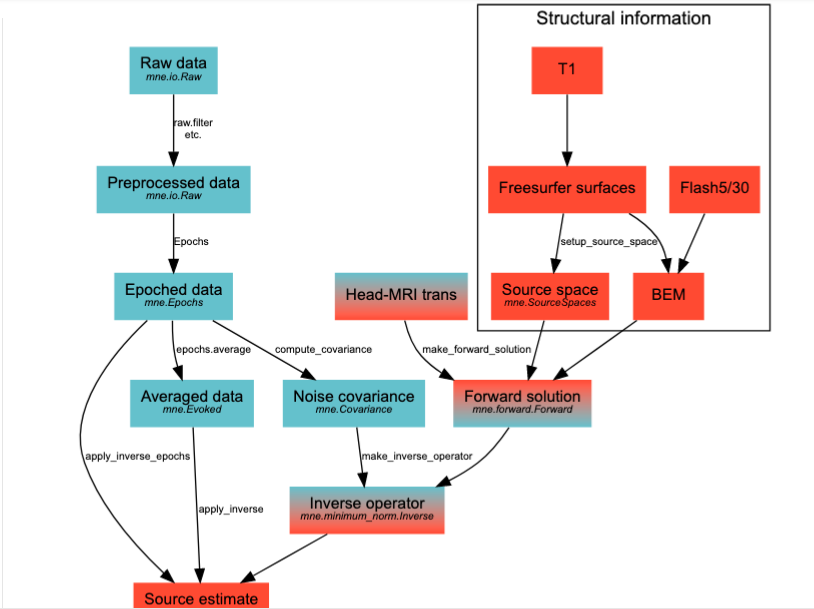
Приведённый ниже код иллюстрирует некоторые шаги, которые необходимы MNE для запуска протокола локализации источника. Опять же пример данных содержит всё необходимое для выполнения этих шагов при условии, что вы перейдёте в нужную директорию. Многие переменные, которые я использовал, можно заменить. Например, последняя переменная, brain, имеет поверхность pilal, но изображение, как в начале этой статьи, может быть сгенерировано, если установить поверхность inflated.
data_path = sample.data_path()
# the raw file containing the channel location + types
raw_fname = data_path + '/MEG/sample/sample_audvis_raw.fif'
# The paths to Freesurfer reconstructions
subjects_dir = data_path + '/subjects'
subject = 'sample'
subjects_dir = pathlib.Path(mne.datasets.sample.data_path()) / 'subjects'
mne.viz.plot_bem(subject='sample', subjects_dir=subjects_dir,
orientation='coronal')
# The transformation file obtained by coregistration
trans = data_path + '/MEG/sample/sample_audvis_raw-trans.fif'
epochs_fname = pathlib.Path('out_data') / 'epochs_cleaned.fif'
info = mne.io.read_info(epochs_fname)
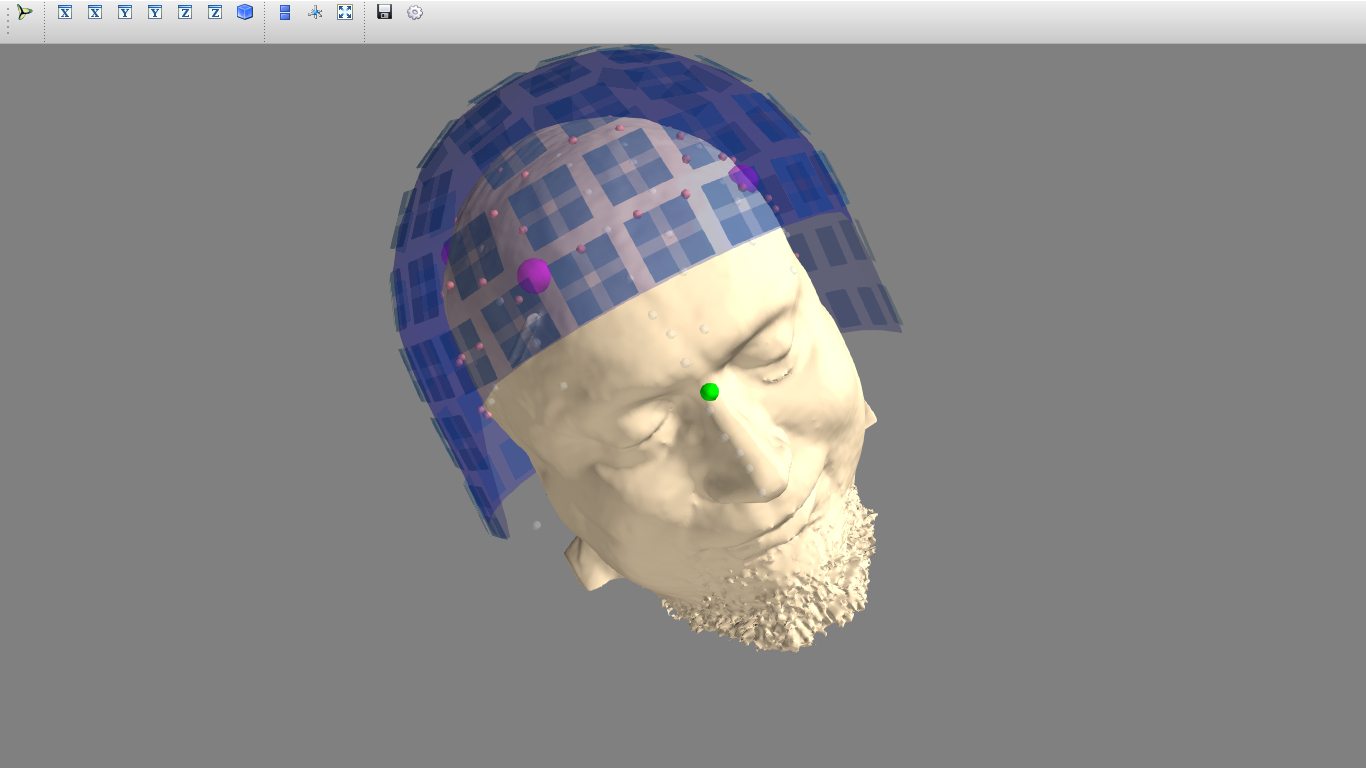
fig = mne.viz.plot_alignment(info=info,
trans=trans,
subject='sample',
dig=True,
subjects_dir=subjects_dir,
verbose=True)
subject = 'sample'
src = mne.setup_source_space(subject=subject,
spacing='oct4', # Use oct6 during an actual analysis!
subjects_dir=subjects_dir,
add_dist=False) # Remove this one during an actual analysis!
mne.viz.plot_alignment(info=info, trans=trans, subject=subject,
src=src, subjects_dir=subjects_dir, dig=True,
surfaces=['head-dense', 'white'], coord_frame='meg')
conductivity = (0.3,) # for single layer – used in MEG
# conductivity = (0.3, 0.006, 0.3) # for three layers – used in EEG
model = mne.make_bem_model(subject=subject, ico=4,
conductivity=conductivity,
subjects_dir=subjects_dir)
bem_sol = mne.make_bem_solution(model)
mne.bem.write_bem_solution('sample_bem.fif', bem_sol, overwrite=True)
fwd = mne.make_forward_solution(info,
trans=trans,
src=src,
bem=bem_sol,
meg=True, # include MEG channels
eeg=False, # exclude EEG channels
mindist=5.0, # ignore sources <= 5mm from inner skull
n_jobs=1) # number of jobs to run in parallel
mne.write_forward_solution('sample_fwd.fif', fwd, overwrite=True)
noise_cov = mne.compute_covariance(epochs, tmax=0.,
method=['shrunk', 'empirical'],
rank='info')
mne.viz.plot_cov(noise_cov, info=info)
epochs.average().plot_white(noise_cov)
inverse_operator = make_inverse_operator(info, fwd, noise_cov,
loose=0.2, depth=0.8)
method = "dSPM"
snr = 3.0
lambda2 = 1.0 / snr ** 2
stc = apply_inverse(epochs['Visual/Right'].average(),
inverse_operator,
lambda2,
method=method,
pick_ori=None)
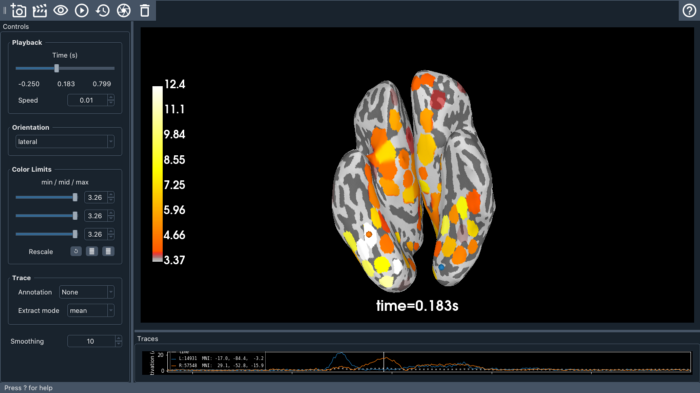
brain = stc.plot(surface='pial',
hemi='both',
subjects_dir=subjects_dir,
time_viewer=True)
# Finished
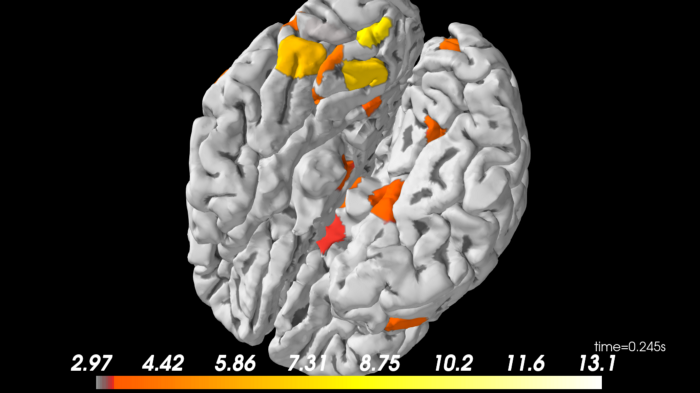
Локализация источника правосторонней визуальной информации дала изображение, показывающее активность в левой затылочной доле и некоторую временную информацию. Также наблюдается некоторая подкорковая активность вблизи таламуса (который передаёт сенсорную информацию.)
Заключение
Ссылки
[1] Gulamhusein A. An introduction to brain-computer interfaces [Internet]. Medium. Geek Culture; 2021 [cited 2021Aug1].
[2] Gramfort A, Luessi M, Larson E, Engemann DA, Strohmeier D, Brodbeck C, et al. MEG and EEG data analysis with MNE-Python [Internet]. Frontiers in neuroscience. Frontiers Media S.A.; 2013 [cited 2021Aug1].
[3] Python homepage¶ [Internet]. MNE. [cited 2021Aug1].
[4] Datasets overview¶ [Internet]. Datasets Overview — MNE 0.24.dev0 documentation. [cited 2021Aug1].
[5] Appelhoff S, Sanderson M, Brooks TL, Vliet Mvan, Quentin R, Holdgraf C, et al. MNE-BIDS: Organizing ELECTROPHYSIOLOGICAL data into the Bids format and facilitating their analysis [Internet]. Journal of Open Source Software. 2019 [cited 2021Aug1].
[6] Holdgraf C, Appelhoff S, Bickel S, Bouchard K, D’Ambrosio S, David O, et al. iEEG-BIDS, extending the brain imaging data Structure specification to human Intracranial electrophysiology [Internet]. Scientific data. Nature Publishing Group UK; 2019 [cited 2021Aug1].
MNE-Python — это невероятный инструмент, но он — только один представитель мира интерфейсов между мозгом и компьютером. Эта статья не учебник, она даёт обзор возможностей MNE и некоторых протоколов, которые можно легко просмотреть при помощи небольшого количества данных. Изучение мозга — также только одно приложение науки о данных из огромного количества возможных приложений. Если вы хотите стать специалистом в области, которая сегодня находит применение почти везде, приходите на наши курсы по Machine Learning, флагманский курс по Data Science или курс по аналитике данных. Также вы можете узнать, как начать карьеру или выйти на новый уровень в других направлениях:

Data Science и Machine Learning
Python, веб-разработка
Мобильная разработка
Java и C#
От основ — в глубину
А также: