Здравствуйте, товарищи! Хочу написать a good story про то, как портировал нейросеть в браузер.
Задача пришла ко мне от моих институтских друзей из ИВМ РАН. Есть некий фронтенд, на который доктор загружает КТ снимок. Доктору предлагается при помощи веб интерфейса выделить сектор с сердцем, который будет передан на сервер, где алгоритмически отсегментируется граф аорты для последующего анализа.
Меня попросили сделать нейросеть для выделения 3d сектора с сердцем, а затрачиваемое время не должно превышать 2-3 секунд.
Гонять весь КТ снимок на сервер только за координатами накладно, т.к. КТ снимок обычно состоит из 600-800 кадров размера 512 * 512 пикселей, поэтому мое предложение о браузерном варианте пришлось кстати.
Рассматривали задачу как задачу сегментации. В качестве модели использовали mobilenetv3. Сначала взяли её версию из пакета segmentation models (https://github.com/qubvel/segmentation_models.pytorch), но её время исполнения было слишком долгим. Потом обнаружили её уменьшенную версию в пакете mediapipe - https://google.github.io/mediapipe/solutions/selfie_segmentation.html, но и её время исполнения было неудовлетворительно. Тогда я подрезал её слои и получилась уже подходящая сетка с 50k параметрами весом всего 210 КБ. Размер изображения и сетки подбирался по скорости обработки всей пачки изображений разом и приемлемой точности. В итоге тренировали на изображениях размера 64 * 64 пикселей.
На рисунке 1 можно увидеть зависимость времени обработки батча от его размера. А на рисунке 2 удельное время обработки кадра из батча.
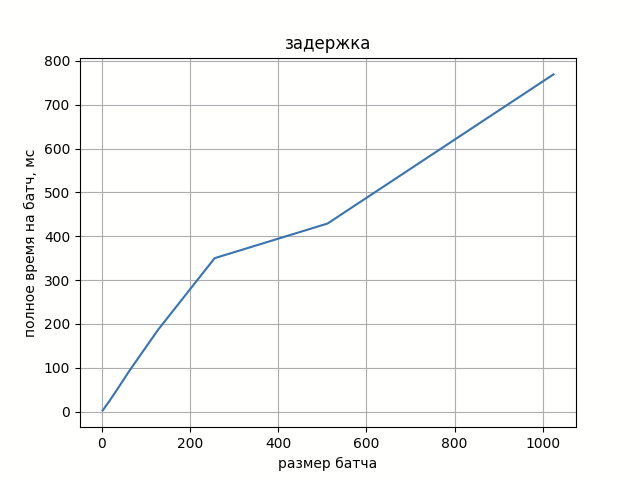
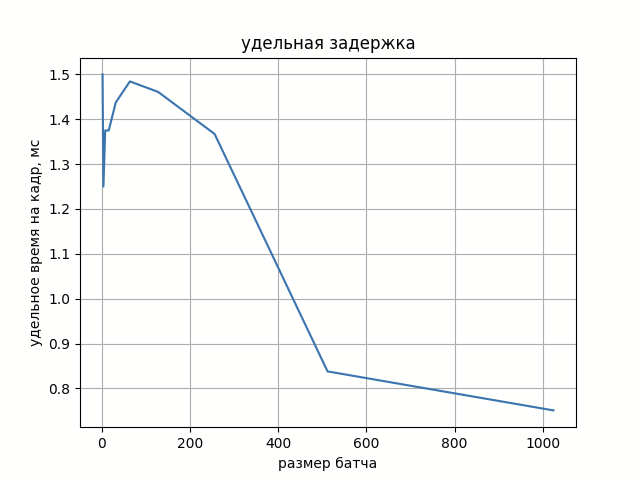
Рост задержки обработки полного батча практически линейно зависит от его размера, а удельная задержка на кадр шумно колеблется вокруг некой константы.
Если добавить время на предобработку фотографий (снижение разрешения с 512 * 512 до 64 * 64), то в сумме получаются желанные 1.5 секунды.
Код был написан на C++, а для портирования под браузер использовался https://emscripten.org/ . Он позволяет довольно просто собрать cmake проект в специальный wasm-модуль, который можно загрузить в JS коде, но все зависимости нужно собрать статически под wasm. В этом проекте потребовались библиотеки opencv и onnxruntime. К счастью, разработчики обеих подготовили для этого инструкции их сборки - https://onnxruntime.ai/docs/build/web.html и https://docs.opencv.org/4.x/d4/da1/tutorial_js_setup.html . Для изменения дефотлного пути установки opencv можно подредактировать https://github.com/opencv/opencv/blob/4.x/platforms/js/build_js.py.
По итогу сборки получаем три файла: main.js, main.wasm, main.data. В файле с расширением .data лежат прикрепленные файлы (в данном случае веса нейросети), с расширением .wasm – скомпилированный C++ код, в main.js – обёртка для загрузки и инстанцирования модуля. При сборке есть возможность ужать всё в 1 js файл, но в этом случае *.data и *.wasm файлы будут преобразованы в base64 и уложены внутрь js файла – а это займет сильно больше места, поэтому мы этим не воспользовались.
Помимо изложенного подхода есть ещё вариант использовать tensorflow.js или фреймворк mediapipe (https://habr.com/ru/post/502440/) с их коллекцией шикарных демо (https://mediapipe.dev/). Или собрать tflite (С++) в wasm (под cmake в старой версии tflite, а свежую –только в проекте под сборщиком bazel). Но мне очень недоставало хорошего репозитория с примером сборки простого пайплайна. Так и появилась данная статья.
Итоговый код и докерфайл для воспроизводимости лежат тут – https://github.com/DmitriyValetov/onnx_wasm_example. У демо есть два режима: первый – обработка 200 целевых фото и вывод детекций, второй – скоростной тест с выводом графиков (ваши показатели могут отличаться от приведенных в настоящей статье, потому что будут зависеть от железа).
В дальнейшем плане имеется идея сделать бенчмарк простой сетки, может даже этой же, но разными фреймфорками: onnxruntime, tflite и mxnet. Openvino пока что не выкатился в wasm, но они поставили эту задачу на google summer of code (https://github.com/openvinotoolkit/openvino/wiki/GoogleSummerOfCode/628ff2fe7f78bc4e07ecd473042cae8374aacba3).