Не последнюю роль в биоинформатике занимает визуализация. Учёные в этой области работают с огромными объёмами информации, которую хорошо бы как-то охватить взглядом и представить в голове. Ярким примером средства визуализации являются браузеры геномов (genome browser), о которых я и хочу рассказать.
Как многие помнят из школьного курса биологии, геном состоит из набора хромосом, а хромосома — это две цепочки, свёрнутые в спираль. Каждая из цепочек содержит последовательность нуклеотидов с четырьмя типами азотистых оснований — аденин (A), гуанин (G), цитозин (C) и тимин (T). По одной цепочке легко определить вторую, если помнить, что аденин соединяется в пару с тимином (Антошка-Тимошка), а гуанин с цитозином (гусь-цыплёнок). Некоторые участки ДНК называются генами, с них считывается РНК, по которой потом кодируются белки. Белки состоят из аминокислот 20 видов (плюс пара экзотических), каждая из которых кодируется по трём нуклеотидам.
Браузер генома — это такая одномерная карта, которая отображает какую-нибудь нуклеотидную последовательность (скажем, хромосому или отдельный ген) с сопутствующей информацией. Информация обычно структурируется в блоки, называемые треками (tracks). К примеру, может быть трек с генами или с отдельными нуклеотидами. Отдельные сущности на треках часто называют фичами (features).
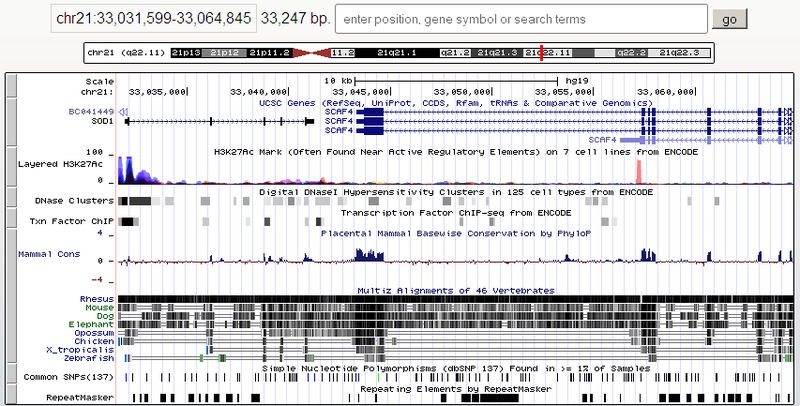
Бывают браузеры геномов, заточенные под маленькие бактериальные геномы, но универсальному браузеру необходимо показывать и длинные хромосомы позвоночных целиком, и отдельные нуклеотиды. Самая длинная хромосома человека (первая) содержит около 250 миллионов пар оснований, то есть масштаб должен меняться примерно в миллион раз. Конечно, в разном масштабе информация отображается по-разному. Например, на картинке выше есть трек с генами UCSC Genes, куда попал ген SOD1 целиком и фрагменты соседних генов. В таком масштабе отображается экзон-интронная структура гена. Экзоны (те части, которые останутся в РНК после сплайсинга и в перспективе закодируют белок) обозначены закрашенными прямоугольниками, а интроны (промежутки между экзонами) — стрелочками, которые показывают направление считывания гена (в данном случае ген SOD1 расположен на прямой нити ДНК, а BC041449 — на обратной). А вот как кусок гена SOD1 выглядит при увеличении:
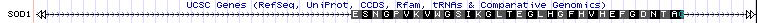
Здесь масштаб позволяет вывести аминокислотную последовательность тех фрагментов гена, которые потом закодируют белок. Каждой аминокислоте соответствует определённая буква латинского алфавита.
Что ещё можно увидеть на браузере генома? В самом детальном масштабе можно увидеть отдельные нуклеотиды, как на прямой, так и на обратной спирали ДНК:

Каждому нуклеотиду соответствует стандартный цвет, поэтому можно весело раскрашивать, даже если сами буквы уже не влезают:

Если ещё немного откатиться, то о нуклеотидном составе можно судить по специальному треку GC content:
Здесь красный цвет означает, что нуклеотидов G и C в данном месте меньше 50%, а синий цвет — больше. Можно подумать, что A, C, G, T — это просто четыре равноправные состояния двухбитовой ячейки, кодирующей генетическую информацию, и доля G и C ни о чём интересном не говорит. Однако пары оснований G-C образуют три водородные связи, а A-T только две. То есть G-C крепче, их труднее разорвать и обогащённость G-C или A-T связями влияет на химические процессы в данном регионе ДНК.
Что ещё интересного можно посмотреть? Обычно имеются треки с геномными вариациями, которые, например, отличают различных людей друг от друга. Часто вариации выражаются в виде точечных мутаций, однонуклеотидных замен (Single-nucleotide polymorphism, SNP). Многие из этих мутаций найдены при сравнении результатов секвенирования геномов разных людей и помещены в специальные базы данных (например, dbSNP):

На приведённом фрагменте не так уж и мало SNP (19 на 356 нуклеотидов — больше 5%). Впрочем, многие из них синонимичны. Так как из 43=64 вариантов трёх нуклеотидов кодируется 20 вариантов белков, некоторые замены не влияют на результирующий белок. Часть замен попадает в некодирующие регионы (например, в интроны), поэтому могут тоже не влиять на результат (но могут и влиять).
Ещё одна интересная штука — это сравнение человеческого генома с геномами других видов. Для этого нетривиальными алгоритмами делают множественное выравнивание геномов и тоже его показывают. На самой верхней картинке поста показано схематично выравнивание человека с макакой-резусом, мышью, собакой, слоном, опоссумом, курицей, лягушкой (Xenopus tropicalis) и рыбкой данио-рерио (zebrafish). Тёмным показаны совпадающие фрагменты. Заметьте, что самые тёмные участки приходятся на кодирующие области генов. На той же картинке есть график консервативности участков среди млекопитающих (Mammal cons), который тоже коррелирует. А вот множественное выравнивание в увеличенном виде:

Минус означает, что нуклеотид есть у человека, но отсутствует у другого вида. Оранжевая вертикальная черта (например, в строчке с собакой между двумя тиминами) — наоборот. Сверху указано количество пропавших нуклеотидов (сами они не приведены). Кодирующий регион приведён в аминокислотном виде, поэтому синонимичных замен не видно. Курица и рыбка, видимо, вообще не имеют похожего региона. Можно убедиться, насколько макака похожа на человека.
На самом дальнем масштабе становится виден кариотип хромосомы:

По кариотипу можно сориентироваться, если помнить, например, в какой полосе находится ваш любимый ген, который вы изучаете. Перекрещивание посередине — это центромера.
Бывает и множество других предопределённых треков. Некоторые браузеры позволяют подгружать треки с веб по специальному DAS-протоколу. Ну и, конечно, геном-браузеры позволяют учёным добавлять свои (для этого есть специальные форматы файлов). Пользовательские треки могут, скажем, показывать области связывания ДНК с конкретным белком (например, с фактором транскрипции), как предсказанные, так и полученные в эксперименте (к примеру, ChIP-Seq). Если вы, к примеру, секвенировали собственный геном, можно загрузить результат и сравнить с референсным и с известными SNP.
Браузеров генома великое множество. Только в Википедии перечислено штук тридцать, а это точно не все. Многие из них специализированы: заточены под определённый организм или определённый тип данных. Из популярных десктопных браузеров можно отметить Integrated Genome Browser и Integrative Genomic Viewer (как видите, с названиями не заморачивались). И тот и другой — Java-приложения, имеется Java Web Start.
Конечно, часто удобнее пользоваться браузером генома на веб. Большинство картинок выше сделаны в UCSC Genome Browser и Ensembl Genome Browser. Оба этих браузера генерируют картинки на сервере. Есть и более современные в техническом плане решения. AnnoJ, например, рендерит картинки на клиенте в canvas, получая от сервера данные в JSON (демонстрация для любимой травки биологов — арабидопсиса). Есть ещё JBrowse. В своём роде он уникален, так как не содержит серверного кода. Данные о треках и последовательностях заранее подготавливаются на сервере в виде статических файлов, которые браузер подгружает по AJAX. Пользовательские файлы обрабатываются через File API.
Идеального браузера геномов не существует. На мой взгляд, основная проблема — это скорость работы. Особенно это заметно на веб, хотя и в десктопных бывают задержки. Некоторые треки при определённых масштабах либо генерируются очень медленно, либо вообще отключаются. Для визуализации приходится перемалывать много информации, которая, возможно, не всегда представлена в оптимальном виде. Поэтому если у кого-то найдётся желание этим заняться, есть все шансы побороть конкурентов.

Как многие помнят из школьного курса биологии, геном состоит из набора хромосом, а хромосома — это две цепочки, свёрнутые в спираль. Каждая из цепочек содержит последовательность нуклеотидов с четырьмя типами азотистых оснований — аденин (A), гуанин (G), цитозин (C) и тимин (T). По одной цепочке легко определить вторую, если помнить, что аденин соединяется в пару с тимином (Антошка-Тимошка), а гуанин с цитозином (гусь-цыплёнок). Некоторые участки ДНК называются генами, с них считывается РНК, по которой потом кодируются белки. Белки состоят из аминокислот 20 видов (плюс пара экзотических), каждая из которых кодируется по трём нуклеотидам.
Браузер генома — это такая одномерная карта, которая отображает какую-нибудь нуклеотидную последовательность (скажем, хромосому или отдельный ген) с сопутствующей информацией. Информация обычно структурируется в блоки, называемые треками (tracks). К примеру, может быть трек с генами или с отдельными нуклеотидами. Отдельные сущности на треках часто называют фичами (features).
Бывают браузеры геномов, заточенные под маленькие бактериальные геномы, но универсальному браузеру необходимо показывать и длинные хромосомы позвоночных целиком, и отдельные нуклеотиды. Самая длинная хромосома человека (первая) содержит около 250 миллионов пар оснований, то есть масштаб должен меняться примерно в миллион раз. Конечно, в разном масштабе информация отображается по-разному. Например, на картинке выше есть трек с генами UCSC Genes, куда попал ген SOD1 целиком и фрагменты соседних генов. В таком масштабе отображается экзон-интронная структура гена. Экзоны (те части, которые останутся в РНК после сплайсинга и в перспективе закодируют белок) обозначены закрашенными прямоугольниками, а интроны (промежутки между экзонами) — стрелочками, которые показывают направление считывания гена (в данном случае ген SOD1 расположен на прямой нити ДНК, а BC041449 — на обратной). А вот как кусок гена SOD1 выглядит при увеличении:
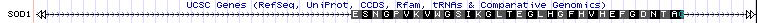
Здесь масштаб позволяет вывести аминокислотную последовательность тех фрагментов гена, которые потом закодируют белок. Каждой аминокислоте соответствует определённая буква латинского алфавита.
Что ещё можно увидеть на браузере генома? В самом детальном масштабе можно увидеть отдельные нуклеотиды, как на прямой, так и на обратной спирали ДНК:

Каждому нуклеотиду соответствует стандартный цвет, поэтому можно весело раскрашивать, даже если сами буквы уже не влезают:

Если ещё немного откатиться, то о нуклеотидном составе можно судить по специальному треку GC content:

Здесь красный цвет означает, что нуклеотидов G и C в данном месте меньше 50%, а синий цвет — больше. Можно подумать, что A, C, G, T — это просто четыре равноправные состояния двухбитовой ячейки, кодирующей генетическую информацию, и доля G и C ни о чём интересном не говорит. Однако пары оснований G-C образуют три водородные связи, а A-T только две. То есть G-C крепче, их труднее разорвать и обогащённость G-C или A-T связями влияет на химические процессы в данном регионе ДНК.
Что ещё интересного можно посмотреть? Обычно имеются треки с геномными вариациями, которые, например, отличают различных людей друг от друга. Часто вариации выражаются в виде точечных мутаций, однонуклеотидных замен (Single-nucleotide polymorphism, SNP). Многие из этих мутаций найдены при сравнении результатов секвенирования геномов разных людей и помещены в специальные базы данных (например, dbSNP):

На приведённом фрагменте не так уж и мало SNP (19 на 356 нуклеотидов — больше 5%). Впрочем, многие из них синонимичны. Так как из 43=64 вариантов трёх нуклеотидов кодируется 20 вариантов белков, некоторые замены не влияют на результирующий белок. Часть замен попадает в некодирующие регионы (например, в интроны), поэтому могут тоже не влиять на результат (но могут и влиять).
Ещё одна интересная штука — это сравнение человеческого генома с геномами других видов. Для этого нетривиальными алгоритмами делают множественное выравнивание геномов и тоже его показывают. На самой верхней картинке поста показано схематично выравнивание человека с макакой-резусом, мышью, собакой, слоном, опоссумом, курицей, лягушкой (Xenopus tropicalis) и рыбкой данио-рерио (zebrafish). Тёмным показаны совпадающие фрагменты. Заметьте, что самые тёмные участки приходятся на кодирующие области генов. На той же картинке есть график консервативности участков среди млекопитающих (Mammal cons), который тоже коррелирует. А вот множественное выравнивание в увеличенном виде:

Минус означает, что нуклеотид есть у человека, но отсутствует у другого вида. Оранжевая вертикальная черта (например, в строчке с собакой между двумя тиминами) — наоборот. Сверху указано количество пропавших нуклеотидов (сами они не приведены). Кодирующий регион приведён в аминокислотном виде, поэтому синонимичных замен не видно. Курица и рыбка, видимо, вообще не имеют похожего региона. Можно убедиться, насколько макака похожа на человека.
На самом дальнем масштабе становится виден кариотип хромосомы:

По кариотипу можно сориентироваться, если помнить, например, в какой полосе находится ваш любимый ген, который вы изучаете. Перекрещивание посередине — это центромера.
Бывает и множество других предопределённых треков. Некоторые браузеры позволяют подгружать треки с веб по специальному DAS-протоколу. Ну и, конечно, геном-браузеры позволяют учёным добавлять свои (для этого есть специальные форматы файлов). Пользовательские треки могут, скажем, показывать области связывания ДНК с конкретным белком (например, с фактором транскрипции), как предсказанные, так и полученные в эксперименте (к примеру, ChIP-Seq). Если вы, к примеру, секвенировали собственный геном, можно загрузить результат и сравнить с референсным и с известными SNP.
Браузеров генома великое множество. Только в Википедии перечислено штук тридцать, а это точно не все. Многие из них специализированы: заточены под определённый организм или определённый тип данных. Из популярных десктопных браузеров можно отметить Integrated Genome Browser и Integrative Genomic Viewer (как видите, с названиями не заморачивались). И тот и другой — Java-приложения, имеется Java Web Start.

Конечно, часто удобнее пользоваться браузером генома на веб. Большинство картинок выше сделаны в UCSC Genome Browser и Ensembl Genome Browser. Оба этих браузера генерируют картинки на сервере. Есть и более современные в техническом плане решения. AnnoJ, например, рендерит картинки на клиенте в canvas, получая от сервера данные в JSON (демонстрация для любимой травки биологов — арабидопсиса). Есть ещё JBrowse. В своём роде он уникален, так как не содержит серверного кода. Данные о треках и последовательностях заранее подготавливаются на сервере в виде статических файлов, которые браузер подгружает по AJAX. Пользовательские файлы обрабатываются через File API.
Идеального браузера геномов не существует. На мой взгляд, основная проблема — это скорость работы. Особенно это заметно на веб, хотя и в десктопных бывают задержки. Некоторые треки при определённых масштабах либо генерируются очень медленно, либо вообще отключаются. Для визуализации приходится перемалывать много информации, которая, возможно, не всегда представлена в оптимальном виде. Поэтому если у кого-то найдётся желание этим заняться, есть все шансы побороть конкурентов.